서 론
재료 및 방법
식물 재료 및 스트레스 처리
RNA 추출 및 QuantSeq 3’ mRNA-sequencing 진행
QuantSeq 3’ mRNA sequencing data 분석
DEGs 분석 및 GO 확인
결과 및 고찰
Alignment 결과
DEGs의 발현 양상 분석
Gene ontology (GO) 분석 및 연관 terms 분류
적 요
서 론
밀, 쌀, 옥수수는 전 세계적으로 가장 중요한 주식이며 일일 열량과 단백질 섭취량의 많은 부분을 차지한다(Kizilgeci et al., 2021). 그중 밀(Triticum aestivum)은 전 세계적으로 곡물 생산 작물 중에 가장 많은 생산량을 가지고 있으며, 인류에 있어 가장 중요한 작물로 여겨진다(Giraldo et al., 2019). 전 세계 인구의 약 36%가 주식으로 밀에 의존하고 있으며 약 20%와 칼로리와 55%의 탄수화물을 제공한다(Karimi & Farshadfar, 2004). 최근 기후변화와 지구 온난화는 수많은 스트레스의 빈도와 정도를 심화시켜 작물의 수확량과 품질에 직접적인 영향을 미치고 있다. 다양한 기후 모델들은 스트레스가 많은 환경 때문에 밀 생산량이 6% 감소할 것으로 예상한다(Asseng et al., 2015).
염분 스트레스는 전 세계 경작지의 약 20%에 영향을 미치며 기후 변화와 인위적 활동으로 인해 지속해서 증가하고 있다. 염분 스트레스는 Na+이온의 흡수를 증가시키고 식물 뿌리 내의 낮은 삼투 포텐셜로 인해 Na+/K+ 비율을 감소시킴으로써 삼투 스트레스 및 이온 독성을 유발한다. 또한, 이러한 이온 불균형은 표적 세포에서 다른 중요한 필수 이온의 흡수 및 수송에 영향을 미치고 중요한 식물 생장 과정과 기능을 방해한다(Arif et al., 2020). 또한 염분은 묘목 형성 저해, 생장 장애, 생식 발달 불량을 발생시키며 궁극적으로 작물 수확량을 감소시킨다(Turan et al., 2009). 염분은 또한 미세 세포 구성 요소를 변경하고, 광합성을 방해하며, 막 구조를 손상시킨다. 더욱이 활성 산소 생산을 증가시키고, 효소 활성을 감소시켜 작물의 생장과 수확량을 제한한다(Hasanuzzaman et al., 2014). 따라서 작물의 염 저항성을 높이려는 노력은 지속 가능한 농업에 매우 중요하며 잠재적으로 작물 수확량을 향상시킨다(Turkan & Demiral, 2009). 식물은 염분 스트레스를 처리하기 위해 이온 항상성, 삼투 항상성, 산화환원 평형, 성장 조절 등을 포함한 특별한 대응 방안들을 가지고 있다. 이러한 방법들은 수많은 염 반응 유전자 발현에 의해 촉진되며, 상응하는 생리적 및 생화학적 변화를 통해 이루어진다. 여기에는 삼투 조절 유전자, 항산화 단백질, 후기 배아 발생(Late embryogenesis abundant) 단백질, 수송체/역수송체, 전사 인자 등이 있다. 예를 들면 ERF 및 WRKY 및 신호 관련 단백질 kinases6를 포함한 구조적 단백질 코딩 유전자 그룹이 포함된다. 위에서 언급한 유전자 간의 상호 작용은 salt overly sensitive (SOS), calcium-dependent protein kinase (CDPK) and the mitogen-activated protein kinase (MAPK) 같은 여러 경로의 기초를 형성한다 (Ludwig et al., 2004; Nakagami et al., 2005; Zhu, 2003). 또한 abscisic acid (ABA), ethylene, salicylic acid 그리고, jasmonic acid 같은 식물 호르몬도 스트레스 신호 및 적응에 중요한 역할을 한다(Fujita et al., 2006; Ma et al., 2006; Xiong & Zhu, 2002).
RNA-Sequencing은 특정 시점에 특정 조직에서 유전자 발현을 연구하는 중요한 접근 방식이다(Wilhelm et al., 2008). RNA transcript profiling은 variant detection, and gene specific expression, genome wide transcript characterization, differential gene expression analysis와 같은 분석들을 할 수 있는 강력한 접근법이다. 이러한 기술들은 염 내성과 같은 복잡한 표현형 형질의 유전적 변이에 대한 더 깊은 이해를 촉진하고 염 스트레스 반응 경로에 대한 이해를 풍부하게 만든다(Han et al., 2015). 여러 RNA sequencing 연구들에서는 보리(Hordeum vulgare L.) (Yousefirad et al., 2020), 고구마(Ipomoea batatas (L.) Lam.) (Luo et al., 2017), 유채(Brassica napus L.) (Yong et al., 2014) 및 애기장대(Arabidopsis thaliana) (Shen et al., 2014)와 같은 다양한 식물에서 염분 스트레스 반응을 조사하였다. 이러한 연구들은 산화-환원 과정과 삼투 보호 대사, 이온 수송, 열 충격 단백질 및 호르몬 신호를 암호화하는 염 내성 유전자의 상당수가 관련되어 있음을 나타낸다.
따라서 염분 내성 관련 유전자 발현을 확인하는 것은 밀 육종 프로그램에서 여전히 중요하며 염분 내성과 관련된 핵심 유전자를 식별하는 데 도움이 된다. 확인된 후보 유전자는 저항성 품종의 개발에 있어 중요하다. 또한 곡물 및 기타 작물에서 미래의 유전 공학 연구를 위한 귀중한 정보를 제공하며 효율적인 적용 육종 계획을 수립하는 데 활용될 것이다. 이번 연구에서는 우리나라에서 가장 많이 재배되는 품종인 금강 밀 품종(Triticum aestivum)과 돌연변이 내염성 밀 돌연변이(2020-s1340, Triticum aestivum)를 통해 염 스트레스 처리 시에 발현되는 전사체를 비교하였다. 차별적으로 발현되는 유전자들을 분석하여 내염성 관련 유전자를 확인하였다. 이러한 결과는 밀의 잎에서 염분 스트레스 반응 메커니즘에 대한 이해를 향상하고 추후 내염성 밀 육종에 있어 유용한 자원이 될 것이다.
재료 및 방법
식물 재료 및 스트레스 처리
국내에서 주로 재배하고 있는 밀 재배종인 금강밀(Triticum aestivum)과 염 저항성을 가지는 돌연변이2020-s1340을 연구 재료로써 사용하였다. 금강밀은 조숙 단간인 관동 75호와 그루밀이 교배된 F1에 연질 조숙인 은파밀을 교배 후 계통 육성하여 선발되었다. 또한, 금강밀은 조숙 고 제분 빵용 장려품종으로써 식물체의 잎은 녹색이고, 간장은 74 cm 내외이고, 이삭의 길이가 짧으며 종실의 색은 백색을 띤다. 2020-s1340 돌연변이는 금강밀에서 gamma radiation을 250Gy 강도로 조사 후 만들어졌다. 만들어진 M9세대 돌연변이는 새만금 토양(염도 2%)에서 스크리닝이 진행되었다. 염도 2% 이상의 환경에서 살아남은 개체를 염 저항성을 가진다고 여기고, 새만금 토양에서 살아남은 식물체에서 얻은 M10세대 종자를 가지고 본 실험을 진행하였다. 종자는 파종 전 오염을 방지하기 위해 70% EtOH와 6% NaOCl을 이용해 소독을 진행하였다. 종자는 growth chamber에서 22°C의 온도로 7일간 발아가 진행되었고 발아 후 같은 온도에서 명(16시간) / 암(8시간) 주기로 유묘기(2엽 출연 및 간장 15~20 cm)에 도달할 때까지 생장시켰다. 식물체들의 염 스트레스 처리를 위해 Hoagland 배양액에 sodium chloride로 200 mM의 농도를 맞추었다. 완성된 배양액을 수조에 깔고 그 위에 마사토로 채워진 포트(깊이 8 cm, 지름 7 cm)를 밑부분만 잠기도록 설치하였다. 그후 식물체 뿌리에 배양액이 삼투작용으로 흡수되도록 하여 뿌리 침수로 인한 습해를 방지하였다. 만들어진 염 처리용 배양액은 12시간마다 전량 교체해 증발로 인한 농도 변화를 예방하였다. 대조군에는 Sodium chloride가 첨가되지 않은 Hoagland 배양액을 사용하였다. 염 스트레스 처리는 48시간 동안 growth chamber에서 진행되었고 온도 및 일조 조건은 이전과 동일하게 사용하였다. 실험이 종료된 후 RNA 추출을 위해 액체 질소를 이용하여 동결시킨 뒤 deep freezer (-80°C)에 보관하였다.
RNA 추출 및 QuantSeq 3’ mRNA-sequencing 진행
RNA 추출을 위해 금강밀과 2020-s1340 두 개의 식물재료에서 각각 염 처리구와 대조구의 잎을 사용하였다. 100 mg잎을 액체 질소와 막자 사발을 이용하여 곱게 갈아준 후 RNeasy plant mini kit (QIAGEN, Germany)을 사용하여 RNA 추출을 진행하였다. RNA의 농도 및 순도는 ND-2000 Spectrophotometer (Thermo Inc., USA)를 사용하여 확인하였고 농도가 100 ng/ul 이상이며 260/280 ratio가 1.8~2.0 범위 안에 있는 RNA만 사용하였다. RNA 품질은 RNA 6000 Nano Chip을 사용하여 Agilent 2100 bioanalyzer (Agilent Technologies, The Netherlands)를 통하여 측정하였으며, RNA Integrity number (RIN) 값이 7 이상을 기준으로 선발했다. Sequencing 진행을 위해 추출된 RNA를 Lexogen QuantSeq 3’ mRNA-Seq Library Prep Kit (Lexogen, Autstria)를 사용하여 제조사의 지시에 따라 cDNA library 제작을 하였다. 각각의 cDNA library 제작을 위해 500 ng의 RNA를 각 샘플 별로 준비하였고 5' 말단에 Illumina 호환 서열을 포함하는 oligo-dT 프라이머를 RNA에 혼합하여 역전사를 수행하였다. 그 다음, RNA 템플릿이 degradation 된 후, 5' 말단에서 Illumina 호환 링커 서열을 포함하는 무작위 프라이머에 의해 두 번째 가닥 합성을 시작했다. 이중 가닥 라이브러리는 모든 반응 성분을 제거하기 위해 마그네틱 비즈를 사용하여 정제되었다. 클러스터 생성에 필요한 완전한 어댑터 시퀀스를 추가하기 위해 라이브러리가 증폭되었다. 그 후, 최종 library는 PCR 구성요소로부터 정제되었다. Sequencing은 QuantSeq 3’ mRNA-sequencing (QuantSeq)을 진행하였다. QuantSeq은 3’UTR 부분만 Sequencing을 진행하는 방식으로 적은 RNA양을 가지고 저렴하면서 빠른 속도로 전사체 발현을 알 수 있다. High-throughput sequencing은 NextSeq 500 (Illumina, Inc., USA)을 사용하여 single-end 75 방식으로 수행되었다(Illumina Inc., USA). 생성된 Sequencing 데이터는 BBduk을 이용하여 Q20 기준으로 필터링을 진행하였다.
QuantSeq 3’ mRNA sequencing data 분석
QuantSeq reads는 Bowtie2를 사용하여 NCBI CH Campala Lr22a Pseudomolecule v5 reference genome (Triticum aestivum)에 정렬되었다. 정렬된 reads는 전사체를 assembly하고, 발현량 확인 및 DEGs확인을 위해 사용되었다. DEGs는 Bedtools에서 coverage를 사용하여 고유 및 다중 정렬의 카운트를 기반으로 결정되었다. Read count데이터는 Bioconductor를 사용하여 quantile normalization 방법에 따라 처리되었다. 유전자 분류는 DAVID (http://david.abcc.ncifcrf.gov/) 및 Medline 데이터베이스(http://www.ncbi.nlm.nih.gov/)에서 수행한 검색을 기반으로 하였다. 분류된 DEGs의 발현 양상을 시각적으로 확인하기 위해 각 개체의 평균 Z-score을 기반으로 MeV를 이용하여 Heatmap 분석을 진행하였다. 또한 작성된 Heatmap을 기반으로 HCL분석을 통해 유전자 간 연관관계 확인을 하였다.
DEGs 분석 및 GO 확인
유의미한 DEGs 선발을 위해 선택해 fold change 2 이상, normalized data (log2) 4 이상, p-value 0.05 이하를 기준으로 필터링하였다. 확인된 DEGs의 annotation은 NCBI Plant Nr Database에 BlastX를 이용하여 cut-off E-value of 1e-5 기준으로 확인하였다. GO functional enrichment 분석은 GO terms에서 DEGs의 기능을 식별하기 위해 수행되었다. DEGs는 AgriGo 2.0 (Go analysis tool kit and database for agriculture community, http://systemsbiology.cau.edu.cn/agriGOv2)을 통해 singular enrichment analysis (SEA)로 기능이 분석되었다.
결과 및 고찰
Alignment 결과
본 연구에서는 sequencing data생산을 위해 우리 밀 품종 금강밀과 염 저항성 돌연변이 2020-s-1340, 두 식물체 잎 조직에서 RNA를 추출해 QuantSeq을 진행하였다. 염 스트레스 처리에 따른 전사체 발현 양상을 살펴보기 위해 실험구에는 식물체에 200 mM NaCl 처리를 하였고, 대조구에는 염 스트레스 처리하지 않았다. 생물학적 2반복 수행을 통해 sequencing을 진행하여 총 8개의 sequencing data를 생산하였다. 그 후 생산된 sequencing data 중, BBduk을 사용하여 phred Quality Score가 Q20 이상인 reads만 사용하였다. 필터링 된 reads는 Bowtie2를 이용하여 reference genome (NCBI CH Campala Lr22a Pseudomolecule v5)에 alignment를 진행하였다. 금강밀 무처리 구의 processed reads는 23,634,438 개가 생산되었고 mapping된 reads는 7,331,269개로 31.08%의 mapping rate를 가진다. 또한 금강밀 염 처리구의 processed reads는 25,306,095개가 생산되었으며 mapping된 reads는 3,975,734개로 25.97%의 mapping rate가 나왔다. 2020-s1340 무 처리구의 processed reads는 23,465,577개이며 mapping된 reads는 7,074,570개, mapping rate는 30.15%를 가진다. 마지막으로 2020-s1340의 염 스트레스 처리구의 processed reads는 23,407,937개이며 mapping된 reads는 5,252,864개로 22.44%의 mapping rate을 가진다(Table 1).
Table 1.
Filtered reads and alignment results of QuantSeq data.
| Sample | Processed read | Mapped read | Mapping rate |
| KCT | 23,591,928 | 7,331,269 | 31.08% |
| K200 | 15,306,095 | 3,975,735 | 25.87% |
| SCT | 23,465,577 | 7,074,570 | 30.15% |
| S200 | 23,407,937 | 5,252,864 | 22.44% |
DEGs의 발현 양상 분석
DEGs의 발현 비교는 금강 200 mM NaCl염처리/금강 무처리(K200/KCT), 2020-s1340 200 mM NaCl염처리/2020- s1340 무처리(S200/SCT), 2020-s1340 200 mM NaCl염처리/금강 200 mM 염처리(S200/K200) 3가지 비교를 통해 확인하였다(Fig. 1A). DEGs 선발은 fold change; 2.00, normalized data (log2); 4.00, p-value (0.005) 기준으로 진행하였다. 그 결과 K200/KCT에서는 상향 발현된 DEGs가 77개, 하향 발현된 DEGs가 37로 총 114개의 DEGs가 확인되었다(Table 2). S200/SCT 비교 조합에서는 45개의 상향 발현된 DEGs, 19개의 하향 발현된 DEGs, 총 64개의 DEGs가 확인되었다(Table 3). 마지막으로 S200/K200 비교 조합에서는 46개의 상향 발현된 DEGs, 58개의 하향 발현된 DEGs 그리고 총 104개의 DEGs가 확인되었다(Table 4). Venn diagram으로 세 비교 조합의 DEGs를 확인한 결과, 세 비교 조합에서 공통적으로 발현되는 DEGs는 확인되지 않았다. S200/K200과 K200/KCT 비교에서는 오직 1개의 상향 발현된 DEGs만 공통적으로 발현되었다. S200/SCT과 S200/K200의 비교를 확인한 결과 6개의 대조 발현 DEGs와 1개의 하향 발현된 DEGs가 공통적으로 존재하였다. K200/KCT와 S200/SCT에서는 공통적으로 2개의 상향 발현 DEGs, 1개의 대조 발현 DEGs, 1개의 하향 발현 DEGs가 확인되었다(Fig. 1B). 각 비교 조합에서 공통적으로 발현된 DEGs가 거의 발견되지 않았다. 이는 금강밀과 저항성 돌연변이의 염 스트레스 처리에 따른 유전자 발현이 서로 독특하게 이루어진다는 것을 나타낸다. 각 비교조합에서 확인된 DEGs를 NCBI Plant Nr database에 BlastX를 이용해 annotation을 진행하였다. Annotation을 통해 기능이 확인된 DEGs를 바탕으로 heatmap을 통해 발현량을 확인하였다(Fig. 2). 분석을 통해 확인된 DEGs중 염 저항성 돌연변이 2020-s1340에서 염 스트레스 처리를 했을 때 상향 발현하는 유전자들 중 염 저항성과 관련된 후보 유전자들이 있을 것으로 예상된다. S200/SCT에서 DEGs 중 blastX 진행 후 기능이 확인된 유전자는 16개로 그 중 CAMPLR22A2D_LOCUS1545는 상향 발현된 유전자로 UDP-glucosyltransferase (UGTs)를 코딩하는 유전자이다. UGTs는 애기장대에서 anthocyanin 축적을 통해 염 스트레스, 가뭄 스트레스, 저온 스트레스에 저항성을 부여한다고 보고된다(Li et al., 2017). CAMPLR22A2D_LOCUS128는 receptor kinase-like protein Xa21을 코딩하는 유전자로 Receptor-like kinase (RLK) protein은 abscisic acid (ABA), salicylic acid, methyl jasmonate, 가뭄, 높은 염도에 의해 유도되며 RLK의 과발현을 갖는 형질 전환 애기장대는 염분 및 가뭄 내성이 개선되고 ABA에 대한 민감도가 증가함을 보인다고 보고된다(Zhao et al., 2013). CAMPLR22A2D_ LOCUS338는 Lectin receptor-like kinases (LecRLKs)을 코딩하며 LecRLKs는 RLK sub-family로 최근 식물 발달 및 비생물적 및 생물학적 스트레스에 대한 반응에서 중요한 역할을 하며 세포 표면을 방어하는 기능을 가진다고 밝혀졌다(Sun et al., 2020). CAMPLR22A2D_LOCUS4890는 cytochrome P450 76T24의 기능을 가지며 Cytochrome P450 (CYP)는 NADPH 및 O2 의존성 하이드록실화 반응에 관여하는 가장 큰 효소군이다. CYP는 또한 증가된 항산화 활성으로 flavonoid와 같은 화합물의 활성을 향상시켜 열악한 환경 조건으로부터 식물을 보호한다(Pandian et al., 2020). CAMPLR22A2D_LOCUS5065는 wall-associated receptor kinase-like 8를 코딩하는 유전자로 wall-associated kinase (WAK) multigene family는 식물체의 다양한 세포 과정과 스트레스 반응에서 중요한 역할을 하는 것으로 알려져 있지만 염 저항성과 관련되어 있는지는 불분명하다. 2020-s1340에서는염처리를 진행했을 때 상향 발현했으나 최근 연구에서는 WAK112를 녹다운 시킨 쌀이 염 스트레스에서 생존율이 향상했다는 연구가 있다. 따라서 이 단백질에 관해서는 추가적인 연구가 필요할 것으로 보인다. CAMPLR22A2D_ LOCUS3327는 6-phosphofructokinase 2의 기능을 가진 유전자로 6-phosphofructokinase는 식물체의 염 스트레스 하에서 인산화를 증가시키는 것으로 보고됐다. 이를 통해 스트레스 내성을 위한 탄소 대사 및 에너지 공급을 향상시킨다(Pan et al., 2021). PFK인코딩 유전자는 벼 묘목에서 관찰되는 것처럼 스트레스 반응을 포함하여 다양한 조직에서 다양한 기능적 역할을 한다는 것이 입증되었다(Mustroph et al., 2013). Alternative oxidase (AOS)는 스트레스 반응에서 아데닐산 조절, 파괴적인 활성 산소(ROS) 형성 축소에 관여할 수 있으며 AOS가 과발현된 애기장대는 낮은 ROS 형성, 개선된 성장률 및 식물체내 Na+ 감소가 이루어진다고 보고 됐다(Smith et al., 2009). 염 저항성 2020-s1340의 염 처리 상황에서 상향 발현된 유전자들은 염 저항성에 관여하는 기능을 가지고 있음을 확인했다. 그러나 몇몇의 유전자의 경우 염 저항성과 연관성이 아직 확인되지 않았다. 2020-s1340에서 상향 발현된 유전자들은 아마도 염 저항성에 관여할 가능성이 높다. 따라서 확인된 유전자들에 대해 염 저항성과 관련된 추가적인 연구가 필요할 것이다.
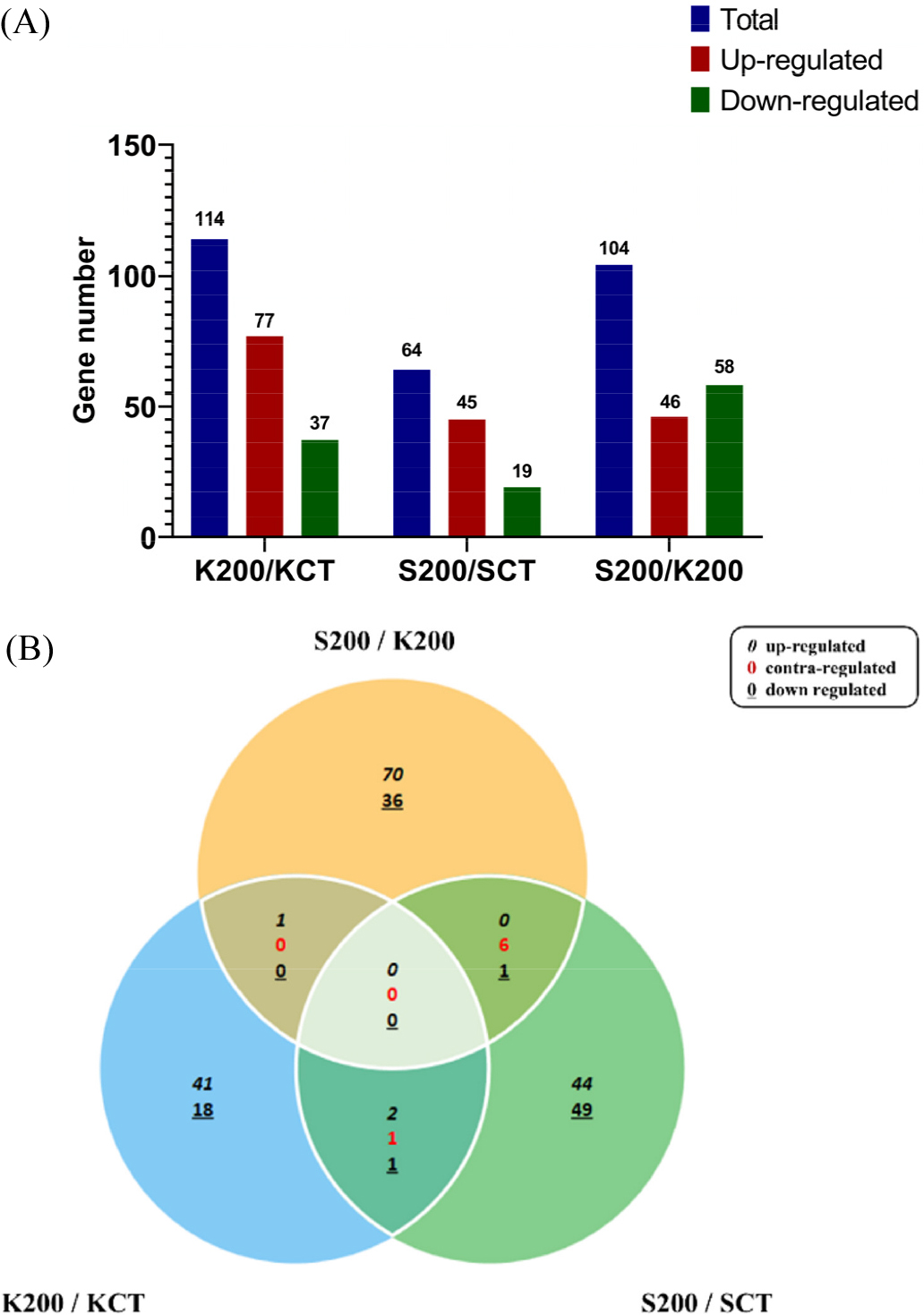
Fig. 1.
(A) Differentially expressed genes (DEGs) analyzed for three comparative combinations (K200/KCT, S200/SCT, S200/K200) of Keumgang and 2020-s1340 in leaves under salt stress condition and control. (B) Venn diagram showing the distribution of overlapping or unique genes. K200; Keumgang 200 mM NaCl, KCT; Keumgang Control, S200; 2020-s1340 200 mM NaCl, SCT; 2020- s1340 Control.
Table 2.
Differentially expressed genes of K200/KCT with NCBI description.
Table 3.
Differentially expressed genes of S200/SCT with NCBI description.
Table 4.
Differentially expressed genes of S200/K200 with NCBI description.
Gene ontology (GO) 분석 및 연관 terms 분류
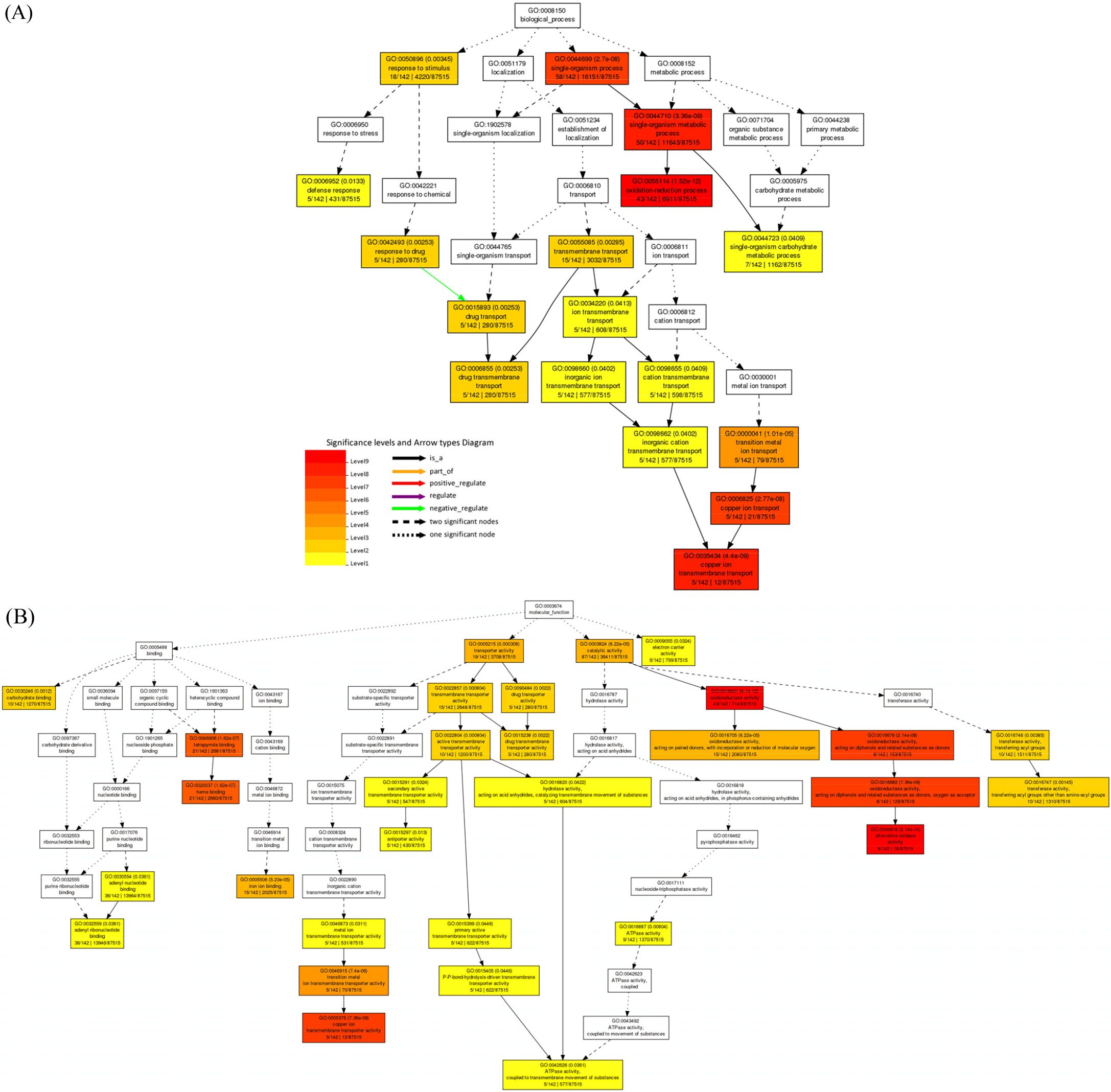
밀 염 저항성에 기초한 분자적인 메커니즘을 밝히기 위해 DEGs를 GO 데이터 베이스를 이용하여 기능을 분류하였다. GO분석은 AgriGo v2.0을 이용해 SEA를 통해 진행하였다. GO의 범주는 크게 ‘Biological process’, ‘Molecular function’, ‘Cellular component’ 3가지로 나뉘어진다. K200/KCT, S200/ SCT, S200/K200 비교 조합에서 각각 염 스트레스 상황에서 차등 발현되는 유전자들의 GO 범주를 확인하였다. K200/KCT DEGs에서는 각각 345개의 GO term들이 연결되었고 S200/ SCT에는 180개, S200/K200에서는 298개의 GO term들이 확인되었다. 3개의 DEGs 모두 biological process에서는 metabolic process (GO:0008152), single-organism process (GO:0044699), cellular process (GO:0009987), primary metabolic process (GO:0044238) 같은 term들이 많이 분포하는 것을 확인하였다. Molecular function에서는 catalytic activity (GO: 0003824), binding (GO:0005488), heterocyclic compound binding (GO:1901363), organic cyclic compound binding (GO:0097159)와 같은 카테고리에 공통적으로 연결이 되었다. Cellular component에서는 membrane (GO:0016020), membrane part (GO:0044425), integral component of membrane (GO: 0016021), cell part (GO:0044464), cell (GO:0005623)와 같은 세포 기초 구성과 관련된 term들의 연결을 확인하였다(Fig. 3). 특히 S200/SCT에서 상향 발현된 유전자들 중 biological process에서 single-organism process (GO:0044699), single- organism metabolic process (GO:0044710), oxidation-reduction process (GO:0055114), copper ion transport (GO:0006825), copper ion transmembrane transport (GO:0035434)와 같은 세부적인 GO term에서 유의성이 높게 확인되었다. 구리는 식물의 일부 단백질 성분으로 엽록체의 안정성을 높이는 것 외에도 광합성에 참여한다. 식물체는 염 스트레스에서 뿌리의 구리 농도를 증가되고 잎의 구리 농도는 감소된다(Guo et al., 2020). 또한 molecular function에서는 alternative oxidase activity (GO:0009916)의 유의성이 높게 나오는데 alternative oxidase (AOX)는 미토콘드리아 기능과 산화 환원 항상성에 관여하여 식물 성장과 스트레스 반응에 필수적인 역할을 하는 것으로 보고됐다. 이전 연구에 따르면 AOX는 비 인산화 대체 호흡경로와 낮은 활성산소 축적을 활성화 시키며 식물체의 염 내성에 관여하는 것으로 확인됐다(Smith et al., 2009). 2020-s1340에서 상향 발현된 DEGs의 유의한 GO term들을 통해 기존 단백질 기능 외에도 염 저항성에 관여하는 기작을 확인 할 수 있었다(Fig. 4). 염분 스트레스는 전 세계적으로 식물의 성장과 발달을 억제하는 요인 중 하나이다. 밀은 세계에서 가장 많이 생산되는 중요한 식량 곡식 작물이다. 밀은 미래 기후변화에 대비해 식량자원으로써 큰 의의가 있다. 그러나 이러한 밀의 중요성에도 불구하고 밀의의 염 스트레스와 관련된 유전적 기전에 대한 연구는 부족하다. 본 연구에서는 염분 스트레스에 대한 저항성과 감수성을 갖는 두 가지 밀 유전자형을 비교 연구하였다. 두 가지 밀 데이터를 가지고 염분 스트레스를 받았을 때 다르게 발현되는 유전자에 대해 확인을 하였다. 이들 유전자를 기반으로 GO 분석과 BlastX를 통해 이들 유전자가 어떤 기능을 하는지 확인했다. 이러한 유전자형의 비교 분석은 스트레스 반응과 관련된 후보 유전자를 제공하고 밀의 염분 내성 메커니즘에 대한 더 나은 이해를 제공할 수 있다. 이 연구는 밀 염분 스트레스의 유전적 조절을 이해하는데 귀중한 자료를 제공할 뿐만 아니라, 염분 스트레스에 대한 작물 내성의 유전적 개선을 위한 실질적인 토대를 마련할 수 있다.
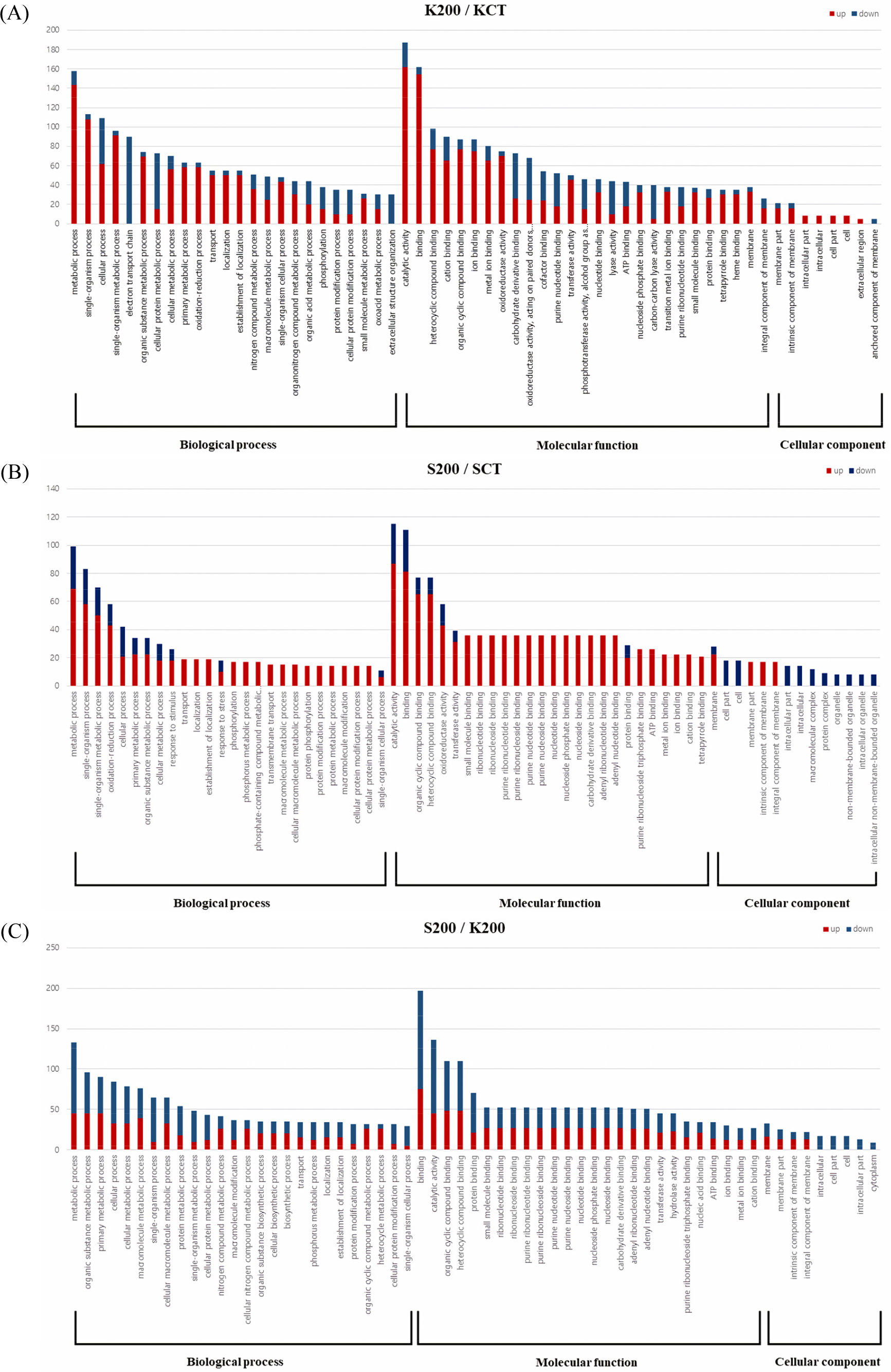
Fig. 3.
Gene ontology (GO) annotations of differentially expressed genes (DEGs) between two wheats genotypes (Keumkang and 2020-s1340) under salt stress. GO category was composed using biological processes, molecular functions, and cellular components. GO term annotation of (A) K200/KCT, (B) S200/SCT, (C) S200/K200.
적 요
1. 본 연구에서는 우리밀 품종인 금강밀과 염 저항성을 가지는 돌연변이 라인 2020-s1340을 재료로 200 mM 염 스트레스 처리에 따른 전사체 발현을 확인하였다. QuantSeq을 통해 23,634,438개의 reads가 생산되었고 7,331,269개의 reads가 mapping됐다.
2. 염 스트레스 상황에서 총 282개의 DEG가 확인이 되었고 이러한 DEGs는 UDP-glucosyltransferase, receptor kinase- like protein, Lectin receptor-like kinases, cytochrome P450등의 단백질들을 코딩하는 유전자들이다. 이러한 DEGs는 염 저항성과 관련된 후보 유전자들이 될 수 있다. 염 저항성과 관련하여 역할이 밝혀지지 않은 유전자들은 추후 연구를 통해 확인이 필요하다.
3. GO연구에서는 DEGs를 세가지 범주로 분류하였으며 대부분 식물체 내 세포 기초 경로와 관련된 GO term들이 주로 되었으며 각각 범주에 있어서 biological process, molecular process에서는 single-organism process (GO: 0044699), single-organism metabolic process (GO:0044710), oxidation-reduction process (GO:0055114), copper ion transport (GO:0006825), copper ion transmembrane transport (GO:0035434), alternative oxidase activity (GO:0009916) GO term들이 유의성이 높게 나타났다.
4. 이러한 QuantSeq의 분석결과는 밀에 관한 염에 의해 발현되는 전사 발현에 대한 이해를 향상시킬 수 있다. 또한 염 스트레스 반응의 복잡한 분자 메커니즘에 대한 좋은 통찰력을 제공하고 염분 스트레스에 대한 작물 내성의 유전적 개선을 위한 실질적인 토대를 마련할 수 있을 것이다.