서 론
재료 및 방법
시험재료 및 콩 발아실험
이소플라본 함량 분석
qRT-PCR에 의한 유전자 발현
통계분석
결과 및 고찰
발아시간 경과에 따른 이소플라본 함량
발아시간 경과에 따른 이소플라본 생합성 관련 유전자 발현
발아시간별 이소플라본 생합성 관련 유전자간 상대적 발현
적 요
서 론
이소플라본은 비스테로이드성 페놀류 식물 화합물로서 콩에 많이 함유되어 있다. 콩의 이소플라본 함량은 환경 및 기후 조건 등 여러 요인에 영향을 받지만 유전적 요소도 중요한 역할을 하는데, Miladinović et al. (2019)은 육종적 기술에 의해 품종의 이소플라본 함량을 높일 수 있다고 하였다. 따라서 최근에는 이소플라본 생합성 관련 유전자에 대한 연구가 활발하게 수행되고 있다(Kim et al., 2021; Lim et al., 2020; Wu et al., 2020; Jia et al., 2019).
콩의 이소플라본은 phenylpropanoid 경로의 한 가지에 의해 합성된다(Fig. 1). 이 경로의 전구체는 L-페닐알라닌이며, 이는 PAL (phenylalanine ammonia lyase), C4H (cinnamate 4-hydroxylase), 4CL (4-coumarate CoA ligase)을 거쳐 Coumaryol CoA로 전환된다. 그리고 플라보노이드 합성을 위한 첫 번째 중요한 효소인 CHS (chalcone synthase)를 시작으로 CHI (chalcone isomerase), IFS (isoflavone synthase), HID (2-hydroxyisoflavone dehydratase)를 거치면서 aglycone (genistein, daidzein)을 생성하며 이는 IF7GT (isoflavone 7-O-uridine diphosphate glycosyltransferase)에 의해 glucoside 형태(genistin, daidzin)로 전환되고 다시 IF7MaT (isoflavone 7-O-glucoside-6-O-malonyltransferase)에 의해 malonyl-glucoside을 생성한다. 한편 isoflavone의 한 종류인 glycitein은 genistein이나 daidzein과는 조금 다른 경로로 합성되는 것으로 알려져 있다(Ahmad eet al., 2017; Gupta et al., 2017; Yuk et al., 2016).
Gutierrez-Gonzalez et al. (2010b)은 isoflavone synthase genes (IFS1 및 IFS2)가 식물체내 이소플라본 축적에 많은 영향을 끼친다고 하였으며, Devi et al. (2020)은 IFS2 유전자가 이소플라본 축적에 핵심 역할을 한다고 하였다. Yuk et al. (2016)은 페닐알라닌에서 이소플라본으로 진행되는 생합성과정에 관여하는 CHS 등 몇 가지 유전자들의 콩잎에서의 발현 정도를 보고하였으며, 외적 스트레스에 의한 이소플라본 합성 관련 유전자들의 발현 양상에 대한 연구 결과가 발표되었다(Lim et al., 2020; Chennupati et al., 2012). 또한 콩 발육단계에 따른 유전자들의 발현량 변화에 대한 연구 결과도 많이 보고되고 있다(Devi et al., 2020; Gupta et al., 2017; Chen et al., 2011). 한편, 이소플라본 생합성 관련 유전자 발현 연구는 형질전환 등 유전자 조작을 통하여 식물체내 관련 유전자의 과발현 혹은 knockdown을 수행함으로써 유전자와 이소플라본 축적과의 관련성에 대한 연구도 보고되었다(Wu et al., 2020; Ahmad et al., 2017; Chu et al., 2017).
이밖에 이소플라본 생합성 관련 유전자 연구는 GWAS (genome-wide association study)를 통한 이소플라본 농도와 관련된 SNP (single nucleotide polymorphisms) 확인(Chu et al., 2017), QTL (quantitative trait loci) 분석(Pei et al., 2018; Gutierrez-Gonzalez et al., 2010b), miRNA 연구(Gupta et al., 2017) 등이 수행되었으며, cytosine methylation에 의한 후성유전학적 조절에 대한 연구도 보고되었다(Gupta et al., 2018).
본 연구는 콩의 발아기간 동안 이소플라본 생합성 관련 몇 가지 유전자들의 발현 양상을 확인하고자 수행되었다.
재료 및 방법
시험재료 및 콩 발아실험
시험재료는 2020년에 수확된 대풍2호와 풍산나물콩 2 품종을 국립식량과학원에서 분양받아 사용하였다. 발아실험은 페이퍼 타올에 콩 종자 각 100립씩을 치상하고 28°C 생육상에서 완전임의 3반복으로 배치한 후 하루에 4번 충분하게 수분을 공급하며 수행하였다. 발아실험 시작 1일, 3일 및 5일째에 공시재료를 시기별로 회수하여 이소플라본 함량 분석에 사용하였다. 단, 발아 5일차 시료는 1일 및 3일차와는 달리 생육이 진전된 상태이었으므로 자엽과 배축을 따로 분리한 후 이소플라본 함량 분석에 사용하였다.
이소플라본 함량 분석
발아시기 별로 수거된 시료를 동결건조 시키고 100 mesh 크기로 분쇄한 후 HPLC (Ultimate3000 HPLC, Thermo Dionex, USA)를 사용하여 이소플라본 함량을 분석하였다. 먼저 시료 0.1 g을 80% 메타놀(30 ml)로 잘 혼합하고 30분간 초음파 처리를 한 후, 다시 1시간 shaking (180 rpm) 시키고 0.2 um PVDF로 여과시킴으로써 전처리를 완료하였다. HPLC 분석은 Inno C-18 칼럼(4.6 × 250, 5 μm, Youngjinbiochrom, Koera)을 사용하여 UV-detector 260 nm 파장에서 수행하였다. 용매는 0.1% formic acid와 acetonitrile을 조합별로 혼합한 후 gradient에 따라 1.0 mL/min 속도로 흘려주었다. 이소플라본 표준물질은 시중에서 구입하여 사용하였는데, aglycone (glycitein, genistein, daidzein)과 배당체(glycitin, genistin, daidzin)는 Extrasynthese사(France), malonyl-glucoside (6’-O-malonyldaidzin, 6’-O-malonylgenistin, 6’-O-malonylglycitin)와 acetyl-glucoside (6’-O-acetyldaidzin, 6’-O-acetylgenistin, 6’-O-acetylglycitin)는 Wako사(Japan)의 제품을 각각 사용하였다.
qRT-PCR에 의한 유전자 발현
Total RNA는 TaKaRa MiniBEST Plant RNA Extraction Kit (Takara Bio Inc.)를 사용하여 추출한 후, ReverTra AceTM qPCR RT Master Mix with gDNA Remover (Toyobo Co., Ltd.)를 사용하여 Reverse transcription을 진행시켰다. cDNA의 합성 이후 NanoDropTM Lite Spectrophotometer (Thermo ScientificTM)을 사용하여 cDNA의 순도를 확인한 후 최종 추출한 모든 cDNA의 농도를 100 ng/μl으로 조정하고 PCR에 사용하였다. Gene primers는 기존의 연구를 참조하여 NCB (National Center for Biotechnology Informations)의 Primer-BLAST에서 확인 후 사용하였으며(Table 1), PCR 수행 후 normalized expression을 확인하기 위한 internal control로는 콩의 SKIP16 유전자를 사용하였으며, relative gene expression은 2-△△Ctmethod (Livak and Schmittgen, 2001) 방법으로 계산하였다. qRT-PCR은 CFX96 Touch Deep Well Real-Time PCR Detection System (Bio-Rad Inc.)을 사용하여 수행되었으며 iQTM SYBR® Green Supermix (Bio-Rad Inc.)를 real-time DNA quantification에 사용하였다. PCR은 initial denaturation 40 사이클 95°C/3분, denaturation 95°C/ 15초, annealing 57°C/30초, extension 72°C/30초로 진행하였다.
Table 1.
Primers used for quantitative real-time PCR.
| Primer | Forward (5’ to 3’) | Reverse (5’ to 3’) | Reference |
| CHS6 | CTCACTTTCCATCTCCTCA | ATCCAAAAGATAGAGTTGTAATCATC | modified Yuk et al. (2016) |
| HID | CACTCTTGCCTTGCTCTAAGTT | CTCAACGGTGTGGTGGTAG | modified Yuk et al. (2016) |
| IF7GT | CCCCACCATTCACCCAAC | TGGCAAGCGTAACTCAAGG | Yuk et al. (2016) |
| IF7MaT | CCCTCTCTTCAAACCTCG | TGGTGGCTTGTTATTCCTAG | Yuk et al. (2016) |
| GmIMaT1 | ATAGAGGAGCAGAATCAGAGCC | CAATCCVACTGTGAACGCAAA | Ahmad et al. (2017) |
| GmIMaT3 | TAACAACCACCGCCGAAAC | ACAAATATGCGCCCACGAT | Ahmad et al. (2017) |
| SKIP16 | GAGCCCAAGACATTGCGAGAG | CGGAAGCGGAAFGAACTGAACC | Hu et al. (2009) |
통계분석
모든 실험은 3반복으로 수행되었으며, 실험결과는 SPSS (Version 17.0, SPSS Institute Inc., Chicago, IL, USA)를 사용하여 통계처리 하였다.
결과 및 고찰
발아시간 경과에 따른 이소플라본 함량
대풍2호와 풍산나물콩을 대상으로 발아시험을 실시한 후 생콩 및 발아 후 1일, 3일, 5일 등 발아시간 경과별로 이소플라본 함량을 분석하였다(Table 2). 국내 콩 육성품종 중 대풍2호는 이소플라본 함량이 높은 반면 풍산나물콩은 낮은 것으로 알려져 있으며(Lee et al., 2018), 또한 대풍2호는 장류콩이지만 풍산나물콩은 콩나물콩으로 사용용도가 서로 다르다. 공시재료인 2 품종 모두 발아시간이 경과함에 따라 이소플라본 함량이 증가하는 양상이었는데 자엽에서의 이소플라본 함량이 3일차에 비해 다소 낮은 것은 5일차 시료에서 배축과 따로 분석되었기 때문이며, 배축에서 이소플라본 함량이 높았던 것은 모든 분석 시료를 일정한 건물중으로 취급하였기에 분석에 사용된 배축의 생체량이 다른 시료들에 비해 많았던 까닭이다.
Table 2.
Mean content (μg g-1, DW) of individual isoflavones during soybean germination.
이소플라본 종류별로는 malonyl-glucoside 함량이 전체의 80% 이상을 차지하였으며, 다음으로 glucoside>aglycone>acetyl-glucoside 순서로 이소플라본 함량이 높았다. 이것은 콩 종자에 함유된 주된 이소플라본은 malonyl-glucoside라는 기존의 보고와 일치한다(Ahmad et al., 2017). 그리고 2 품종 모두 acetyl-glucoside 함량은 거의 분석되지 않았으며 acetyl-daidzin 만이 미미한 함량을 나타내었다. Aglycone 함량에서 glycitein은 생콩에서 거의 분석되지 않았으나 발아시간이 경과함에 따라 함량이 높아졌으며 특히 배축에서는 그 함량이 높아져 대풍2호의 경우 genistein 보다 많이 축적되었다.
자엽의 총 이소플라본 함량은 생콩 및 발아 1일차, 3일차 등 보다 낮았으며 이소플라본 종류별 함량은 생콩 및 발아 1일차, 3일차 등과 비슷한 양상으로 축적되어지지만 특별히 aglycone 함량은 3일차 등의 시료들 보다 자엽에서 높았다. 배축에서는 이미 언급한 glycitein 경우 뿐만 아니라 풍산나물콩에서는 glycitin 함량이 genistin 보다 높았으며, malonyl-glucoside의 축적 양상이 자엽을 비롯한 다른 시료들과 다르게 나타났다. 따라서 발아기간 동안 이소플라본 종류별 축적 정도는 조직에 따라 다르며, 그 차이는 공시품종 간에도 약간 다르게 나타난다는 것을 알 수 있었다. 콩 종자에서 이소플라본 축적은 환경의 영향을 받으며 자엽이 배축보다 환경에 더 민감하다는 연구결과(Berger et al., 2008)가 있으며, 콩에서 이소플라본 함량은 조직에 따라서 다르게 축적된다는 연구 결과도 보고된 바 있다(Ahmad et al., 2017; Gutierrez-Gonzalez et al., 2010). 이와 같은 결과를 통해 기존의 연구(Lim et al., 2020; Jia et al., 2019; Lee et al., 2018)등과 같이 콩 발아 시 이소플라본 함량은 유전적 요인에도 영향 받는다는 것을 알 수 있었으며, Gutierrez-Gonzalez et al. (2010)은 콩에서 이소플라본 함량은 유전적 요인, 환경적 요인뿐만 아니라 유전적 요인과 환경적 요인의 상호작용도 관여한다고 하였다.
발아시간 경과에 따른 이소플라본 생합성 관련 유전자 발현
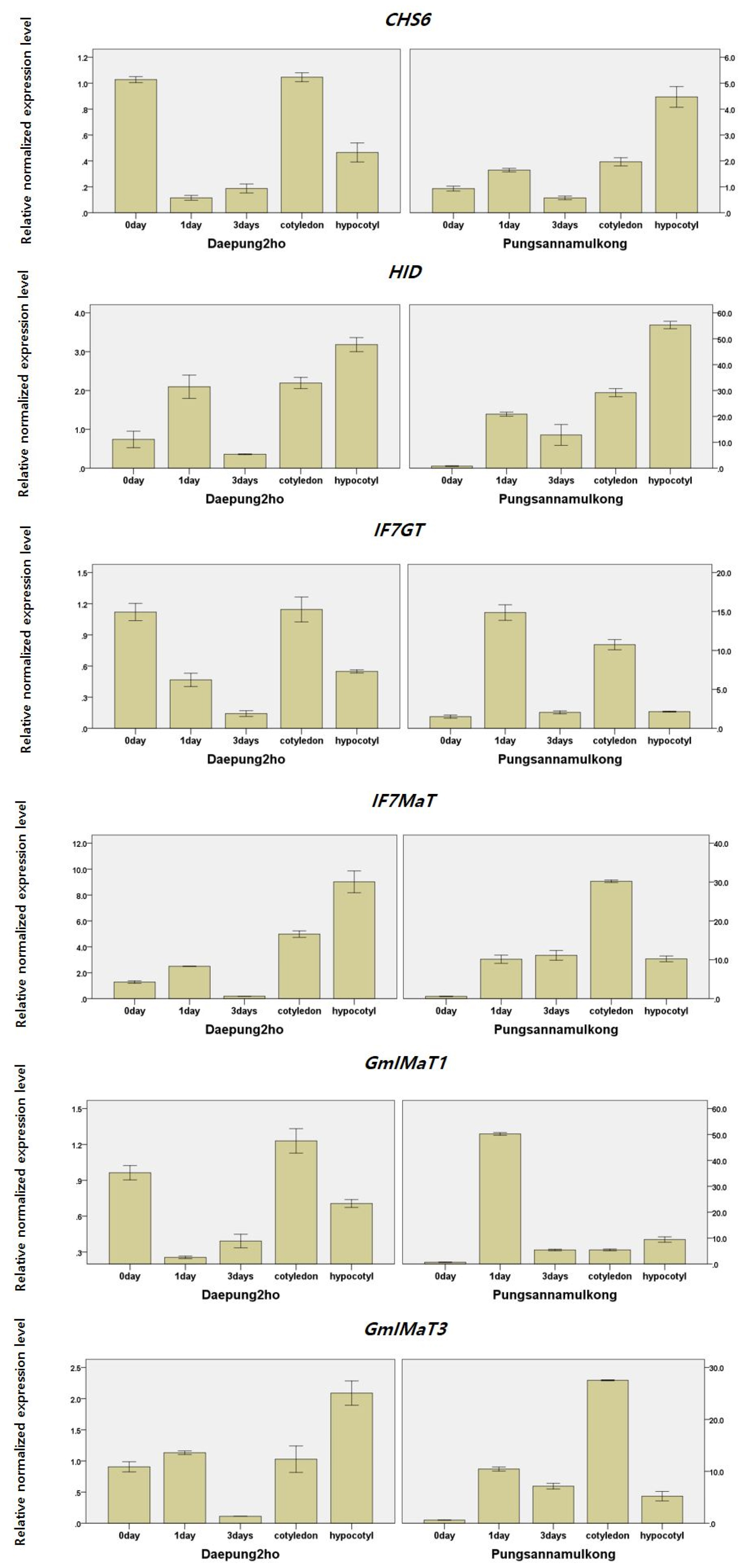
콩의 이소플라본은 phenylalanine으로 부터 시작되어 다양한 효소에 의해 이소플라본 종류별로 단계적으로 발달되어 합성된다. 본 연구에서는 플라보노이드 합성을 위한 첫 번째 중요한 효소인 CHS를 발현시키는 유전자 중 하나인 CHS6, aglycone (genistein, daidzein) 생성에 관여하는 HID, glucoside 형태(genistin, daidzin)로 전환시키는 IF7GT, malonyl-glucoside를 생성하는 IF7MaT 및 isoflavone malonyltransferase 효소 생성에 관련된 유전자 중 일부인 GmIMaT1과 GmIMaT3 등 총 6 개 유전자가 콩 발아기간 동안 어떻게 발현되는지를 확인하였다. 그런데 glycitein은 genistein이나 daidzein과는 조금 다른 경로로 합성되는 것으로 알려져 있어 본 연구에서는 CHS6만 glycitein 합성과 관련되어 사용된 유전자라 할 수 있겠다(Fig. 1). 한편, qRT-PCR 수행시 내재유전자의 역할이 중요한데 그동안 많은 연구에서 콩의 내재유전자로 액틴과 튜불린을 주로 사용하였지만 본 연구에서는 Fbox 단백질 형성에 관여하는 SKIP16을 사용하였다. Hu et al. (2009)은 14개 reference genes들의 안정성 평가 결과 SKIP16, UKN1 및 UKN2 유전자들이 가장 안정하다고 하였으며, Wan et al. (2017)도 콩에서 qRT-PCR 분석시 사용되는 9개의 reference genes들의 안정성 평가에서 생육시기별 분석에는 Fbox와 G6PD 유전자가 최고의 안정성을 보인다고 하였다.
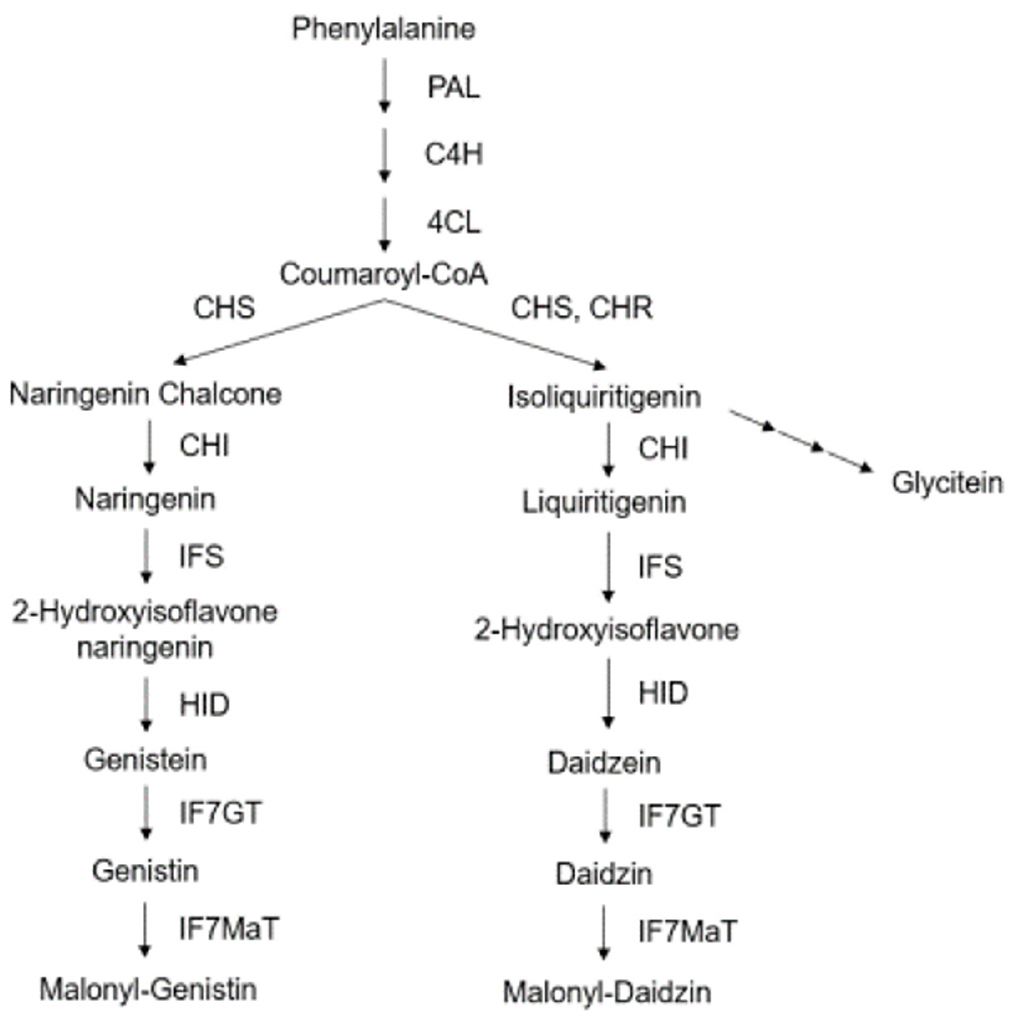
Fig. 1.
Schematic representation of the phenylpropanoid pathway for isoflavone biosynthesis (adapted from Yuk et al., 2016). Abbreviations: PAL (phenylalanine ammonia lyase), C4H (cinnamic acid 4-hydroxylase), 4CL (4-coumarate: CoA ligase), CHS (chalcone synthase), CHR (chalcone reductase), CHI (chalcone isomerase), IFS (isoflavone synthase), HID (2-hydroxyisoflavone dehydratase), IF7GT (isoflavone 7-O-uridine diphosphate glycosyltransferase), IF7MaT (isoflavone 7-O-glucoside-6-O-malonyltransferase).
대풍2호와 풍산나물콩의 발아기간 동안 이소플라본 합성 유전자들의 발현 양상은 품종별로 다르게 나타났으며, 유전자간 발현 양상도 서로간 일정한 경향이 없이 다르게 나타났다(Fig. 2). CHS6의 경우, 대풍2호에서는 생콩에서 발현량이 높았다가 발아 1일 및 3일차에는 오히려 줄어들고 자엽에서는 높게 배축에서는 낮게 발현되었다. 풍산나물콩에서는 발아 3일차에 발현량이 낮아졌으나 발아기간 경과시 대부분 발현량이 높아져 대풍2호와 다른 양상을 나타내었다. CHS6 외 다른 유전자들도 일반적으로 대풍2호에서는 유전자의 발현량이 높고 낮음을 반복하였지만 풍산나물콩에서는 대체로 발아시간 경과에 따라 유전자들의 발현량이 높아지는 경향이었다. Table 2에서 보는 바와 같이 이소플라본 함량은 2 품종 모두 발아시간 경과에 따라 높아지는 경향이었으므로 본 연구 결과로써는 이소플라본 함량과 유전자의 발현량 사이에 일정한 상관을 찾아 볼 수 없었다. Gutierrez-Gonzalez et al. (2010)는 콩 성숙기 동안 한발처리는 이소플라본 생합성 유전자들의 발현량에 영향을 미치지만 유전자의 발현량과 이소플라본 함량과는 상관이 없다고 하였다. 이는 이소플라본 합성에 관련되는 효소 생성에 관여하는 유전자가 다수 존재하므로 1개 유전자의 발현량만으로는 모든 것을 설명하기 어렵기 때문인 것으로 판단된다. 즉, 본 연구에서 CHS6의 발현량을 확인하였지만 CHS 효소 생성 관련 CHS 유전자는 적어도 9개 존재하는 것으로 알려져 있다(Yuk et al., 2016). 이밖에도 효소로 작용하는 단백질들의 수명, 효소의 안정성 등도 영향을 미치는 것으로 판단되는 바 이에 관한 연구는 추후 더 정밀하게 진행되어야 할 것이다. 한편, 이소플라본 생합성 유전자의 발현량은 genotype 별로 차이가 있는 것으로 보고되었다(Kim et al., 2021; Gupta et al., 2017; Chen et al., 2011).
HID 유전자는 대풍2호와 풍산나물콩 모두 발아 3일차를 제외하고는 발아시간 경과에 따라 유전자의 발현량이 높아졌다. IF7GT의 경우 대풍2호에서는 CHS6와 같이 높았다가 낮아지고 다시 높아졌다가 낮아지는 모습을 보였지만, 풍산나물콩에서는 생콩에 비해 1일차에 15배 이상 높아졌다가 3일차에는 발현량이 떨어지고 자엽에서 다시 10배 이상 높아졌다가 배축에서 낮아지는 모습을 보였다. Gutierrez-Gonzalez et al. (2010b)은 R5, R6, R7 등 콩 성숙기별로 종자를 수확한 후 이소플라본 생합성 유전자들의 발현 양상을 분석한 결과, 유전자의 발현 양상에 따라 첫째, 초기에 낮았다가 높아지고 다시 낮아지는 그룹, 둘째, 초기에 높았다가 시간 경과에 따라 낮아지는 그룹, 셋째, 초기에 낮았다가 점차 높아지는 그룹 등 3가지로 구분할 수 있다고 하였으며, Kim et al. (2021)도 생육시기별로 유전자들의 발현양상은 다양하다고 한 바 있어 본 연구의 결과를 뒷받침 하는 것으로 판단된다. IF7MaT, GmIMaT1 및 GmIMaT3 유전자들도 발현량이 발아시간, 풍종간에 다른 양상을 보였다. 한편, NCBI의 Primer-BLAST에서 확인한 결과 IF7MaT와 GmIMaT3는 같은 단백질을 생성하는 것으로 나와 있는데 본 연구에서도 유전자의 발현양상이 거의 같은 경향임을 알 수 있었다.
발아시간별 이소플라본 생합성 관련 유전자간 상대적 발현
이소플라본 생합성의 초기 단계에 관여하는 CHS6 발현량을 기준으로 했을 때 이후 생합성 되는 유전자들의 CHS6 대비 상대적 발현량을 발아시기 별로 분석하였다(Table 3). 대풍2호의 경우 모든 유전자가 발아시간 경과에 상관없이 CHS6보다 상대적 발현량이 높았다. 특히 HID의 발현량이 높았는데 발아 1일차 및 배축에서 CHS6에 비해 각각 59배 및 17배 높았다. 발아시간 별로는 발아 1일차가 다른 발아일수에 비해 유전자들의 발현량이 높은 것으로 나타났으며, 배축에서도 HID가 17배, IF7MaT가 12배 높았다. 이것은 Table 2에서 보여주는 것처럼 대풍2호의 이소플라본 함량이 1일차에 높아지고 다시 배축에서 높아지는 것과 연관이 있으리라 판단된다. 그러나 풍산나물콩에서는 대풍2호와 다른 양상을 보였다. 발아 전 생콩에서 CHS6의 발현량이 가장 높았던 까닭에 다른 유전자들의 상대적 발현량은 기준을 넘지 못했으며, 발아기간이 경과함에 따라 CHS6의 발현량을 넘어서는 유전자가 있는 반면 발현량이 모자라는 유전자도 있었다. 이 역시 풍산나물콩의 이소플라본 함량이 발아시간 경과에도 불구하고 크게 높아지지 않는 것과 연관이 있으리라 생각되나 이미 언급하였다시피 효소 생성에 관련되는 유전자가 다수 존재하는 등 여러 요인이 관여될 수 있으므로 추후 더 많은 연구가 필요할 것이다. 이외에 Fig. 2에서 보는 바와 같이 CHS6의 발현량이 발아일수에 따라 변동 폭이 크게 나타나므로 CHS6 발현량을 기준으로 다른 유전자의 발현량을 비교 평가하는 것에 대한 적절성 여부가 제기될 수 있으리라 사료된다. 그러나 앞에서 언급하였다시피 본 논문에서는 CHS6가 초기에 생합성되는 유전자이므로 이후 생합성되는 다른 유전자 발현량과의 연관성을 찾아보고자 하였을 따름이며, 시기에 따라 유전자의 발현량이 다르게 나타나는 것은 여러 논문에서도 확인되고 있으므로 이 또한 추후 보완연구가 필요하리라 판단된다. Gutierrez-Gonzalez et al. (2010a)는 phenylpropanoid 경로의 첫 번째 단계인 PAL 유전자 발현량을 기준으로 R6시기에 이소플라본 생합성 유전자들의 상대적 발현량을 분석한 결과, 생합성 관련 유전자들은 PAL과 대동소이 하거나 높게 나타났으나 한발 스트레스를 받은 콩에서는 거의 대부분 유전자들의 상대적 발현량이 PAL에 비해 1/3까지 떨어진다고 하였다. 따라서 이소플라본 생합성 유전자들의 발현량은 환경적 요소 및 유전적 요인의 영향을 같이 받는다고 하였다.
Table 3.
Relative expression levels of isoflavone biosynthetic genes compared with the CHS6 gene during soybean germination.
콩에서 이소플라본 함량은 조직 및 기관에 따라 다르게 나타난다. Fig. 3은 발아 5일차 시료의 자엽과 배축에서 CHS6을 기준으로 다른 유전자들의 상대적 발현량을 비교한 결과인데 품종 뿐만 아니라 조직에서도 발현량이 다르게 나타남을 확인할 수 있었다. 다만 자엽에서는 GmIMaT1을 제외한 다른 유전자들의 발현량이 대풍2호와 풍산나물콩에서 비슷하게 나타났다. 배축에서는 HID 발현량이 2 품종 모두 높게 나타난 것을 제외하고는 일치되는 경향을 찾을 수 없었다. Chen et al. (2011)은 이소플라본 생합성 유전자들의 발현량은 품종간 차이가 있으며 조직 및 기관에 따라서도 차이가 있으나 조직특이적이지 않고 발현정도에 일정한 경향이 있다고 하여 본 논문과는 약간의 차이가 있었다. 그러나 Kim et al. (2021)등 많은 논문에서 유전자의 발현량은 콩 발달 단계에서 일정한 경향이 없다고 한 바 있으며, Lim et al. (2020)은 콩 발아기간 중 UV 처리 등을 한 후 발아 7일차에 이소플라본 생합성 유전자들의 발현량을 분석한 결과, 처리 별로 유전자들의 발현량은 달라진다고 하였다.
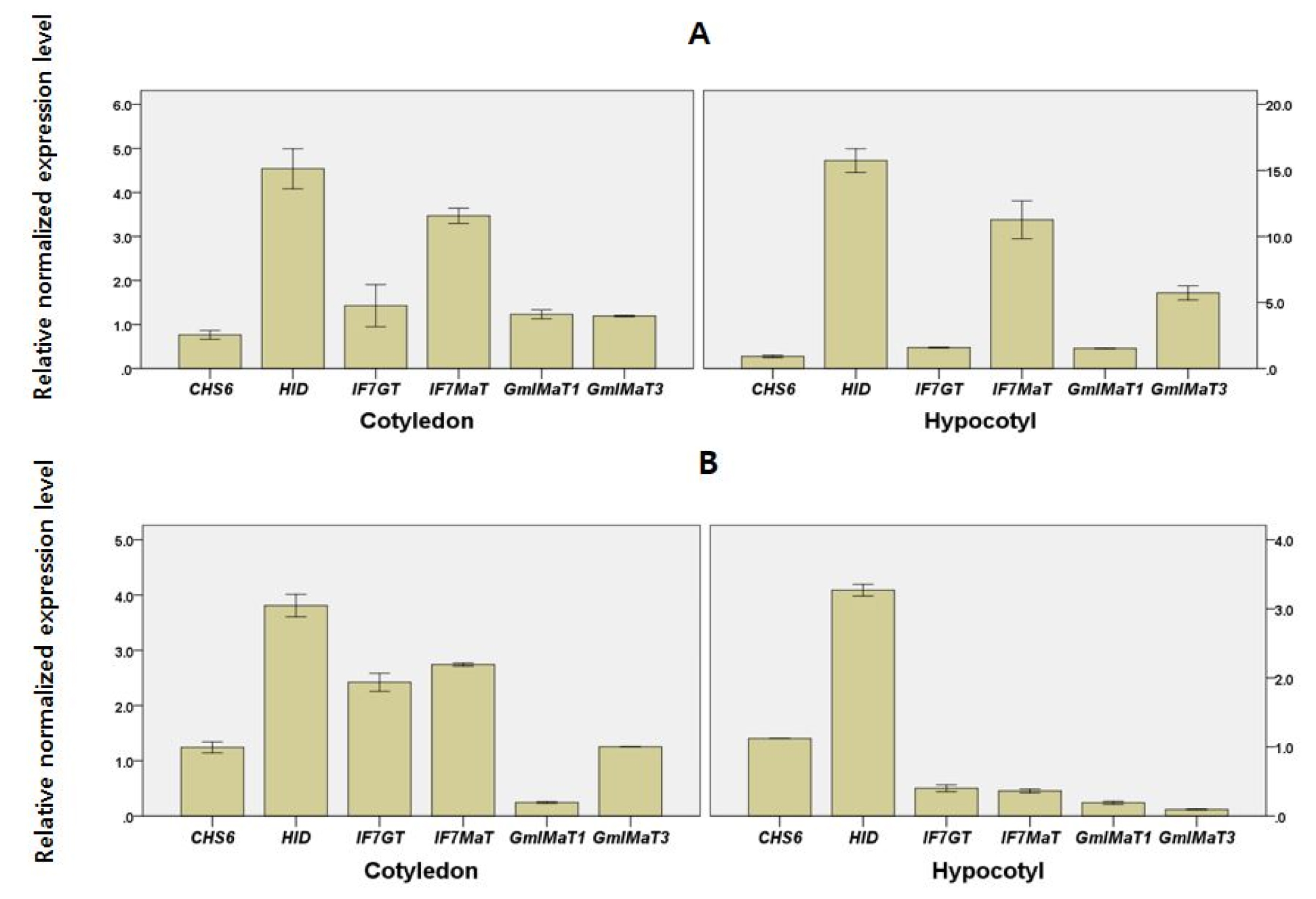
Fig. 3.
Relative expression levels of isoflavone biosynthetic genes in the cotyledon and hypocotyl at 5 days after germination in soybeans cultivars ‘Daepung2ho’ (A) and ‘Pungsannamulkong’ (B). Relative transcript levels were analyzed using qRT-PCR. Gene expression levels were normalized to the expression of the internal control SKIP16 and calculated based on the CHS6 value.
이상과 같이 콩 발아기간 동안 이소플라본 생합성 관련 유전자들의 발현량을 분석한 결과 유전자 발현량은 품종 간 차이가 있으며 발아기간 경과에 따라서도 다르게 나타나나 일정한 경향은 찾을 수 없었다. 이는 관련 효소 생성에 다수의 유전자가 관여한다는 사실 외에도 전사조절의 문제, 효소 활성의 변화 등도 영향을 끼치는 것으로 판단된다. 한편, 콩 발아시기 경과에 따라 이소플라본 함량이 높아지는 것처럼 이소플라본 생합성 관련 유전자들의 발현량도 발아시간 경과에 따라 높아지므로 이소플라본 축적 정도와 유전자들의 생합성 정도와는 상관이 있는 것으로 판단되나 각 유전자의 발현량이 시기별로 다르게 나타나는 까닭에 12종인 개별 이소플라본 함량과의 상관은 구명할 수 없었다. 최근에는 이소플라본 생합성 유전자에 관한 연구도 GWAS (Wu et al., 2020; Chu et al., 2017), miRNA (Gupta et al., 2017) 등을 활용하는 등 여러 시도가 진행되고 있으므로 추후 지금보다 더 명확한 연구결과가 발표되리라 사료된다.
적 요
이소플라본 함량이 높으며 장류콩인 대풍2호와 이소플라본 함량이 낮으며 나물콩인 풍산나물콩을 재료로 하여 콩 발아기간 동안 이소플라본 생합성 관련 유전자 6종(CHS6, HID, IF7GT, IF7MaT, GmIMaT1 및 GmIMaT3)의 발현량을 qRT-PCR로 분석하였다.
1. 공시재료 모두 발아기간 경과에 따라 이소플라본 함량이 높아졌으며, 총 이소플라본 함량 중 malonyl-glucosides 함량이 80% 이상을 차지하여 제일 높았으며 acetyl-glucosides는 거의 분석되지 않았다. 한편, 자엽과 배축에서 이소플라본 축적 정도가 각각 다르게 나타났으며 개별 이소플라본 함량에서도 차이가 있었다.
2. 이소플라본 생합성 관련 유전자들은 콩 발아시기 경과에 따라 발현량이 높아져 이소플라본 축적 정도와 상관이 있는 것으로 판단되나 유전자들의 발현량이 시기별로 각각 달라 개별 이소플라본 함량과 유전자간 뚜렷한 상관은 찾을 수 없었다.
3. HID 유전자는 대풍2호와 풍산나물콩 모두 발아 3일차를 제외하고는 발아시간 경과에 따라 유전자의 발현량이 높아졌으나 다른 유전자들의 상대적 발현량은 품종 간 차이가 있었으며, 또한 발아시간 경과에 따른 유전자의 발현 양상은 유전자별로 각각 달랐다.
4. 발아시간 별로 유전자들의 상대적 발현량을 비교한 결과 품종 간 차이가 있었으며 자엽과 배축에서도 상대적 발현량이 다르게 나타났다. 자엽에서는 GmIMaT1을 제외한 다른 유전자들의 발현량이 대풍2호와 풍산나물콩에서 비슷하게 나타난 반면, 배축에서는 HID 발현량이 2 품종 모두 높게 나타났으나 다른 유전자들은 유전자 발현량에 일정한 경향이 없었다.