서 론
재료 및 방법
시험 재료 및 육성
내염성 선발 적정 농도 선정
내염성 계통 선발
Saltol QTL 유래 확인을 위한 PCR 및 염기서열 분석
SKC1 유전자 발현 양상 분석
데이터 분석
결과 및 고찰
내염성 계통 기내 선발을 위한 염 농도 선정
내염성 계통 표현형 분석
내염성 계통 내 Saltol QTL 분자마커 분석
내염성 계통 내 SKC1 발현 양상 분석
적 요
서 론
벼는 가장 중요한 작물로써 전세계 인구의 1/3 이상이 주식으로 이용한다. 하지만 염분, 열, 가뭄 및 침수와 같은 다양한 환경스트레스는 벼를 비롯한 다양한 작물의 생육 및 생산성을 감소시킨다(Welch et al., 2010; Cramer, 2011). 다양한 환경적 요인 중 전 세계 총 면적의 약 6%와 관개 면적의 1/3이 직접 또는 이차 염분의 영향을 받고 있으며, 경작 가능 토양의 50%가 염의 영향을 받을 것으로 추정된다(Mahajan & Tuteja, 2005; Rahman et al., 2017).
우리나라는 국토 절대 면적이 협소하고 산지가 65%를 차지하며, 최근 농경지의 도시화 및 산업화에 따른 농지전용으로 토지 수요를 충족하기 위한 간척지 개발이 불가피한 실정이다. 우리나라 간척지 면적은 약 13만 5,100 ha로서 국내 총 경지면적의 9%를 차지한다(Lee et al., 2015). 간척지 토양은 일반 농지와 비교하여 염분의 함량이 높아 작물생육에 적합하지 않으며, 이로 인하여 적정수준의 작물 생산성을 유지하는데 어려움이 있다(Lee et al., 2003). 또한 최근 중국 및 인도 등의 경제발전에 따른 식량 및 사료용 곡물의 수요 증가와 바이오에너지 원료를 생산하기 위한 곡물 수요가 급증함에 따라 국제 곡물의 수급여건이 악화되고 있는 실정이다. 따라서 간척지내 재배 가능한 내염성 작물의 개발은 간척지의 효율적 이용 및 곡물의 원활한 수급을 위한 효과적인 방안이라 할 수 있다.
벼는 염 스트레스에 민감하지만 간척지와 같은 염류토양에서 재배하기 좋은 작물 중 하나이다(Mori & Kinoshita, 1987). 벼는 유묘와 생식생장기 모두에서 염 스트레스에 취약하며, 주로 삼투 불균형, 막 불안정화 그리고 광합성 효율 저해와 같은 요인에 의해 피해를 입는다(Munns & Tester, 2008; Vinod et al., 2013). 염 스트레스는 유묘기에서 작물의 생장을 지연시키고 생식생장기에서 수율을 감소시킴으로써 작물 손실을 유발한다(Moradi & Ismail, 2007). 그럼에도 불구하고, 유묘기에서의 내염성은 생식생장기로 이어지는 초기 생장을 촉진함으로써 염분이 발생하기 쉬운 지역에서 작물 수율을 유지할 수 있다(Hoang et al., 2016).
벼의 내염성은 기공의 변화, 이온의 전달, 흡수 및 구획화, 삼투방지제의 생합성 및 축적, 항산화 효소의 조절 등 다양한 형태학적, 생리학적 및 대사 반응(Hanin et al., 2016; Reddy et al., 2017; Chen et al., 2017)과 복합적 요인에 의해 결정되기 때문에 농업현장에서 직접 선발은 반복성과 정밀도에서 불리하다. 따라서 내염성 선발은 실험실 내 유묘기 단계에서 생식생장기 및 영양생장기와 비교하여 신속성과 규모성에서 유리하다(Bhowmik et al., 2007). 또한 내염성 작물 개발을 위한 전통적인 농작물 개량 기술이 한계에 도달함에 따라 스트레스 반응 유전자의 식별을 위한 분자마커 시스템 개발 등 새로운 선발 방법이 요구되고 있다(Telem et al., 2016). 그러므로 높은 수준의 내염성 품종을 개발하기 위해서는 내염성 메커니즘의 기본이 되는 유전자 조절에 대한 이해가 필요하다(Bizimana et al., 2017).
벼에서는 내염성 재래 품종인 Pokkali의 1번 염색체에서 내염성 관련 quantitative trait loci (QTL) Saltol이 확인되었으며, Nona Bokara에서 Saltol 영역에 위치한 SKC1 (OsHKT1;5)이 확인되었다(Bonilla et al., 2002; Ren et al., 2005; Thomson et al., 2010). QTL Saltol은 염 스트레스 시 Na+/K+ 항상성을 조절함으로써 내염성을 부여한다(Ren et al., 2005; Thomson et al., 2010). 애기장대에서 AtHKT1은 뿌리에 Na+ 이온의 축적과 목질부로부터 Na+ 이온의 회수를 조절함으로써 내염성을 증가시킨다(Davenport et al., 2007). 또한 밀에서 Na+ 수송체인 TaHKT1;5-D는 줄기에 Na+을 축적시킴으로써 내염성을 증가시킨다(Byrt et al., 2014).
최근 Marker-Aided Selection (MAS)은 내염성을 포함하여 다양한 특성들을 확인하는 가장 효과적인 방법으로 널리 사용되고 있다(Thomson et al., 2010; Vu et al., 2012; Vinod et al., 2013). Saltol QTL은 내염성을 전달하기 위한 유전자좌로 가장 잘 알려져 있으며, 많은 나라에서 IR29/ Pokkali 교배 계통에서 유래한 내염성 RIL 계통인 FL478 (IR 66946-3R-178-1-1)을 이용하여 다양한 육종 프로그램에 사용되고 있다(Singh et al., 2017). 내염성 교배 계통 내 Saltol QTL 전이를 확인하기 위해서 RM3412, AP3206 및 RM8094와 같은 Simple Sequence Repeat (SSR) marker들이 사용되고 있다(Huyen et al., 2012; Thomson & Ismail, 2016; Babu et al., 2017).
본 연구는 높은 바이오매스 수율을 보이는 자포니카 목양벼와 내염성 인디카 계통 IR64-Saltol 간 교배로 IR64-Saltol 내 Saltol QTL을 목양으로 전이하여 내염성은 유지하면서 우수한 농업 형질을 보이는 교배 계통을 선발하여 간척지와 같은 불량토양에서 재배 가능한 내염성 자포니카 품종 육성을 위해 기내 선발과 분자마커 분석을 수행하였다.
재료 및 방법
시험 재료 및 육성
본 연구에서는 농촌진흥청 중부작물부에서 육성한 자포니카 우량 품종 목양과 내염성 인디카 품종 IR64-Saltol의 교배 계통을 사용하였다. 두 품종 간 단교배 F1을 육성한 후 이 F1에 다시 목양벼를 여교배하여 BC1F1을 육성하였다. 이 후 목양벼와 두번의 여교배를 실시하여 BC3F1을 육성하였다. BC3F1 계통은 single seed descent (SSD) 법으로 F4 세대까지 세대 진전하였고, 그 후에는 계통육종법으로 세대를 진전하여 고정 계통을 육성하였다.
내염성 선발 적정 농도 선정
내염성 계통 기내 선발을 위한 유묘검정체계 확립을 위해 목양과 IR64-Saltol 종자를 이용하여 다양한 농도의 염 조건에서 신초 길이, 근장 및 생체중을 조사하였다. 신초 길이와 생체중을 조사하기 위해 각각의 종자를 50°C에서 이틀간 휴면타파 후 종피를 제거하여 70% ethanol에 30초간, 6% sodium hypochlorite 용액에 20분간 침지하여 소독하였다. 살균된 종자는 멸균된 증류수로 4~5회 세척하여 물기를 제거한 후 0, 20, 30, 50, 70, 및 90 mM NaCl을 함유한 1/2 MS 배지(Murashige & Skoog, 1962; 3% sucrose, 0.7% agar)에 치상하여 기내 배양실 25°C, 광주기 16/8시간(광/암) 조건에서 일주일간 배양 후 신초 길이 및 생체중을 조사하였다. 근장을 조사하기 위해 종자 소독 후 0, 20, 30, 50, 70, 및 90 mM NaCl 용액이 포함된 seed germination pouches (PhytoTC, Beijing, China)에 종자 치상 후 기내 배양실에서 일주일간 수직 배양 하였다. 신초 길이, 근장 및 생체중은 각각의 개체에 대하여 조사하여 평균값을 사용하였다. 내염성 선발 적정 농도는 IR64-Saltol과 비교하여 목양의 신초 길이, 근장 및 생체중이 현저하게 감소하는 농도를 내염성 선발 농도로 결정하였다.
내염성 계통 선발
내염성 계통을 선발하기 위해서 목양과 IR64-Saltol 교배 54계통 224개체 종자의 종피를 제거한 후 70% ethanol에 30초간, 6% sodium hypochlorite 용액에 20분간 침지하여 소독하였다. 살균된 종자는 멸균된 증류수로 세척 후 50 mM NaCl이 함유된 1/2 MS 배지에 치상하여 기내 배양실에서 25°C, 광주기 16/8시간(광/암) 조건에서 일주일간 배양한 후 표준 품종 목양을 기준으로 신초 길이와 생체중을 조사하였고, 소독한 종자를 seed germination pouch에 일주일간 수직 배양 후 근장을 조사하여 염 스트레스 조건에서 생육이 양호한 개체를 1차 선발하였다. 이렇게 1차 선발된 개체들을 각각 계통으로 하여 유리온실에서 BC3F6 세대까지 양성하고 일부 계통의 경우 세대를 더 진전하여 고정 계통을 육성하였다. 이후 각각의 고정 계통 종자를 이용하여 목양 대비 신초 길이, 근장 및 생체중이 우수한 계통들을 기내 선발 방법을 통해 2차 선발하였다.
Saltol QTL 유래 확인을 위한 PCR 및 염기서열 분석
선발된 내염성 계통 내 Saltol QTL 이입 유형을 확인하기 위해 polymerase chain reaction (PCR) 및 염기서열 분석을 수행하였다. PCR과 염기서열 분석을 위한 DNA는 기내 배양실 조건에서 일주일간 배양한 Mogyang, IR64-Saltol 및 내염성 선발 8계통에서 DNeasy Plant Mini Kit (Qiagen, Hilden, NRW, DE)를 이용하여 추출하였다. 추출된 DNA를 이용하여 기존에 보고된 벼 1번 염색체의 Saltol QTL 내 위치한 12개 마커 RM1287, AP3206, SKC1, SKC10, RM3412b, RM10748, PECT4, RM493, RM10793, SalT1, RM562 및 RM7075를 이용하여 PCR을 수행하였다. 이후 PCR 산물을 정제하여 LabChip GX Touch 24 Nucleic Acid Analyzer (PerkinElmer, Waltham, MA, USA)를 이용하여 Saltol QTL 이입 유형을 분석하였다. 이후 정제된 PCR 산물을 이용하여 염기서열 분석을 수행한 후 Saltol QTL 유래를 분석하였다. PCR 및 염기서열분석은 Table 3에 표기된 프라이머를 사용하여 수행하였다.
SKC1 유전자 발현 양상 분석
SKC1 유전자 발현 분석은 부모 계통 목양과 내염성 선발 8계통을 이용하여 수행하였다. 염 스트레스 처리를 위해 각 계통 종자를 1/2 MS 배지에 치상하여 기내 배양실에서 일주일간 배양한 후 200 mM NaCl을 0, 1, 6, 그리고 15 시간 처리하였다. 이후 액체질소를 이용하여 식물을 곱게 분쇄하였고, RNeasy Plant Mini Kit (Qiagen, Hilden, NRW, DE)를 이용하여 RNA를 추출하였다. RNA 농도는 NanoVueTM Plus Spectrophotometers (GE healthcare, Illinois, USA)를 이용하여 정량하였고, RNA to cDNA EcoDryTM Premix (Oligo dT) Kit (Takara, Moriyama, JP)를 사용하여 cDNA를 합성하였다. 유전자 발현 분석에 사용한 프라이머는 NCBI Primer-BLAST를 이용하여 제작하였다. SKC1 증폭을 위해 Fw: 5'-CCTGCCACCTTACACCACTT-3', Rev: 5'-GTAG TTGATGGGGTCGTCGG-3', ACTIN2 증폭을 위해 Fw: 5'- gtatgccag tggtcgtacc-3', Rev: 5'-TCCTGTGCACAATGGA TGG-3'를 사용하였다. Quantitative RT-PCR은 TB Green® Premix Ex TaqTM II (Tli RNaseH Plus) (Takara, Moriyama, JP)를 사용하여 수행하였다.
데이터 분석
모든 시험은 3반복 수행하여 결과를 획득하였으며, 각각의 결과에 대한 통계 분석은 SPSS 통계 패키지(SPSS Inc., Illinois, USA)를 이용하였다. 통계적 검증은 분산 분석과 Student t-test를 수행하였으며, 사후 검정은 터키 검증(Tukey’s HSD)을 사용하여 p 값이 0.05 이하인 경우를 유의한 차이가 있는 것으로 판단하였다.
결과 및 고찰
내염성 계통 기내 선발을 위한 염 농도 선정
본 연구에서는 내염성 증진 계통 선발을 위해 농촌진흥청 중부작물부에서 육성한 자포니카 우량 품종 목양과 내염성 인디카 품종 IR64-Saltol 교배 계통을 사용하였다. 내염성 계통 선발을 위해서는 다양한 외부 환경조건을 배제할 수 있는 in vitro 실험이 요구되며, 식물 생육 및 선발을 위한 염분의 적정 농도 설정이 필요하다. 벼는 유묘기가 염분에 가장 민감한 시기로 유묘기에서 내염성 계통을 선발할 경우 선발 효과가 있음이 보고되었다(Xie et al., 2000). 내염성 선발 적정 농도를 설정하기 위해 목양과 IR64-Saltol 종자를 각기 다른 염 농도를 처리한 후 조사한 결과, 염분농도가 점차 증가함에 따라 신초 길이, 근장 및 생체중이 모두 감소하였다(Table 1). 염 스트레스가 없는 조건에서 IR64-Saltol은 목양 대비 증가된 신초 길이와 근장을 보인 반면, 생체중은 목양에 비해 감소하였다. 신초 길이, 근장 및 생체중은 20 mM과 30 mM NaCl 처리 시 목양과 IR64-Saltol의 생육에 큰 영향을 미치지 않은 반면, 50 mM NaCl 처리 시 목양의 신초 길이는 정상 조건(67.7 ± 3.6 mm) 대비 25.8% 감소한 50.2 ± 3.3 mm를 보였으며, 이는 정상 조건(73.9 ± 3.9 mm) 대비 5.2% (64.2 ± 5.1 mm) 감소한 IR64-Saltol에 비해 현저한 생육 저해를 보였다. 또한 목양의 근장은 정상 조건(60.0 ± 3.9 mm) 대비 34.3% (39.4 ± 6.4 mm) 감소하였고, 생체중은 정상 조건(9.7 ± 0.4 mg) 대비 30.9% (6.7 ± 0.3 mg) 감소하였다. 반면, IR64-Saltol의 근장은 정상 조건(81.2 ± 5.1 mm) 대비 8.9% (78.6 ± 5.1 mm) 감소하였고, 생체중은 정상 조건(8.3 ± 0.8 mg)에 대비 16.9% (6.9 ± 0.5 mg) 감소하였다. 또한 70 mM과 90 mM NaCl 처리시에는 IR64-Saltol 대비 목양에서 신초 길이, 근장 및 생체중이 심각한 생육 저해를 보였다. 따라서내염성이 증진된 계통을 선발하기 위한 염 농도로 표준 품종 목양에서 신초 길이, 근장 및 생체중이 현저하게 감소하는 50 mM NaCl 농도를 내염성 선발의 적정 농도로 선정하였다.
Table 1.
Phenotypic analyses of Mogyang and IR64-Saltol rice cultivars in response to various concentrations of NaCl.
내염성 계통 표현형 분석
목양과 IR64-Saltol 교배 54계통 224개체에서 내염성 계통 선발을 위해 기내 선발 방법을 통해 염 스트레스 처리 시 표준 품종 목양 대비 신초 길이, 근장 및 생체중 등 생육이 양호한 계통을 선발하였다. 목양에 50 mM NaCl 처리 시 신초 길이, 근장 그리고 생체중이 정상 조건 대비 약 37%, 44% 및 30%가 감소함을 확인하였고, 이와 비교하여 신초 길이나 근장 혹은 생체중이 양호한 5계통(767883, 767885, 767949, 767986, 767989)을 선발하였다(Table 2). 선발된 5계통은 정상 조건에서 목양보다 증가된 신초 길이를 보였으나, 염 처리 후 767885 계통은 신초 길이가 약 39% 감소하여 목양과 차이를 보이지 않았고, 767883, 767949, 및 767986 3계통은 목양보다 생육이 양호하였다. 또한 767949와 767989 계통은 정상 조건에서는 목양과 큰 차이를 보이지 않으나 염 처리 후 생육이 양호함을 확인하였다. 염 처리시 신초 길이는 767883, 767949, 767986과 767989 4계통에서 양호하였고, 근장은 선발된 5계통 모두 목양 대비 양호하였다. 또한 생체중은 767883, 767885, 767949와 767986 4계통이 양호하였다. 선발된 5계통 중 767986 계통은 목양과 비교하여 신초 길이, 근장 그리고 생체중 모두가 가장우수한 생육 특성을 보였다.
Table 2.
Phenotypic analyses of salt-tolerant lines in response to 50 mM NaCl.
Asterisks denote significant differences between Mogyang and other lines in the NaCl-treated samples, as determined by one-way ANOVA followed by post hoc Tukey's HSD tests (*, p < 0.05; **, p < 0.01; ***, p < 0.001). The numbers in parentheses indicates the reduced growth rate of NaCl-treated samples compared to normal conditions.
이와 같이 생육상태를 대조구와 비교하여 선발하는 방법은 벼(Zhang et al., 1995)와 밀(Noori & McNeilly, 2000) 등 여러 작물에서 이용되었다. 이를 통해 본 실험에서 육성한 계통은 염에 대한 저항성을 보이는 것으로 확인되었다.
내염성 계통 내 Saltol QTL 분자마커 분석
Saltol QTL은 유묘기의 내염성과 Na+/K+ 함량 조절에 관여하며 이를 확인할 수 있는 다양한 마커들이 알려져 있다(Huyen et al., 2012; Thomson & Ismail, 2016; Babu et al., 2017; Fig. 2A). Saltol QTL 이입 유형을 확인하고자 다양한 마커 중 Saltol QTL 전체를 포함하는 19개 마커(Thomson et al., 2010)를 이용하여 PCR을 수행하였고, 목양과 IR64-Saltol 계통 간 PCR 산물의 크기를 구분할 수 있는 12개 마커를 선택하여 PCR과 염기서열 분석을 수행하였다(Fig. 1, Table 3). 분자마커 분석을 통해 선발된 내염성 8계통에서 다양한 Saltol QTL 이입 유형을 확인하였다. 내염성 5계통 중 767885와 767989 계통은 목양과 IR64-Saltol 유래 Saltol QTL이 혼재되어 있고, 767883, 767949와 797986 계통은 IR64-Saltol 유래 Saltol QTL이 이입되어 있음을 확인하였다. 목양과 IR64-Saltol 유래 Saltol QTL 혼재 이입 계통 중 767885와 767989 계통은 목양 4개 부위와 IR64-Saltol 8개 부위가 이입되어 있음을 확인하였다. IR64-Saltol의 QTL만 이입된 767883, 767949와 767986 계통은 목양과 IR64-Saltol 유래 QTL이 혼재되어 있는 767989 계통과 비교하여 목양 대비 신초 길이, 근장 그리고 생체중이 모두 증가함을 확인하였다. 이러한 결과로 미루어보아, 각각의 계통에서 나타나는 내염성은 IR64-Saltol 내 존재하는 Saltol QTL의 이입 유형과 관련이 있을 것으로 판단된다(Table 4).
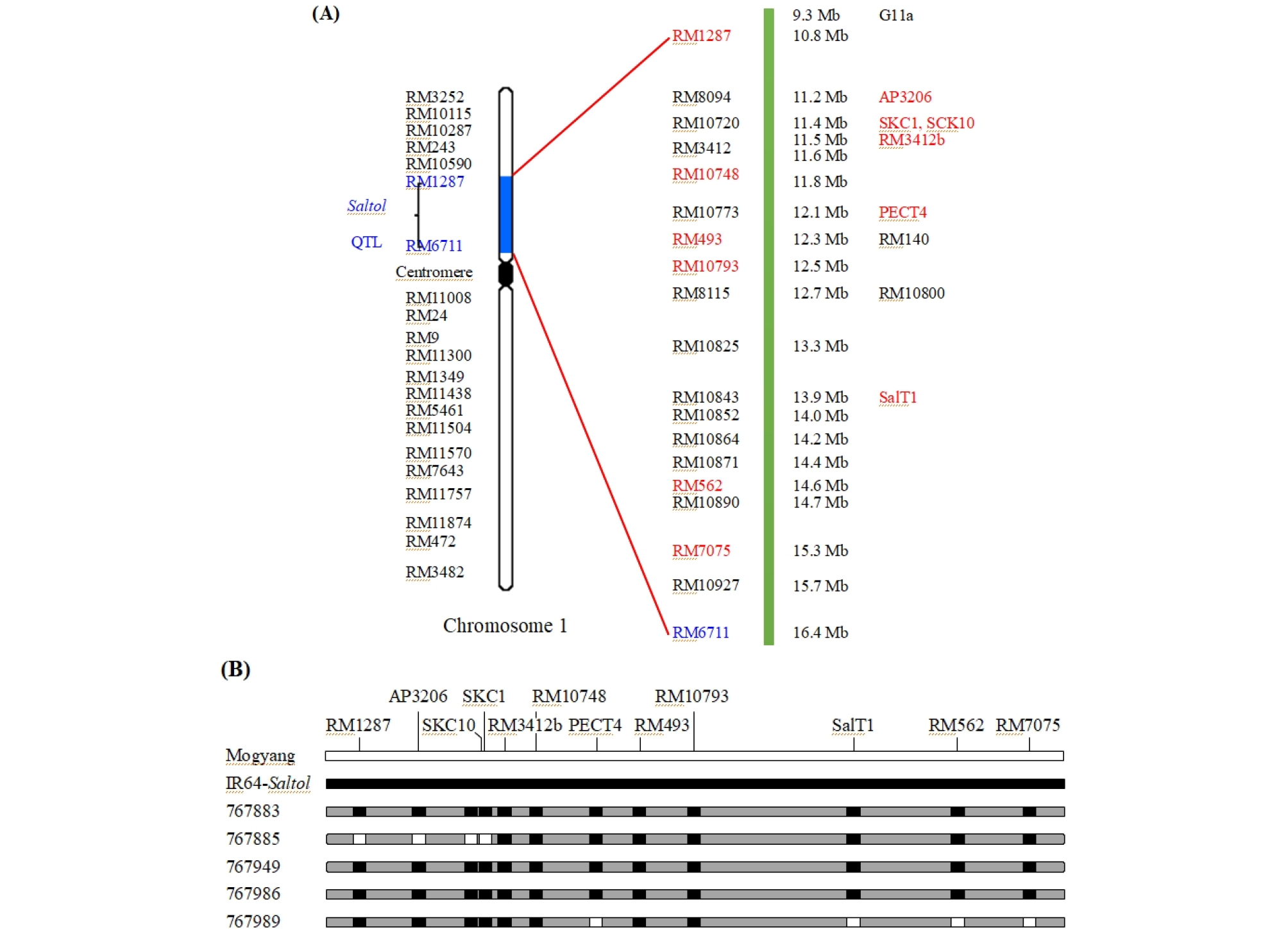
Fig. 1.
Molecular markers analysis for the introgression of Saltol QTL in salt-tolerant lines. (A) Location of Saltol QTL markers on rice chromosome 1. Red letters indicate the markers used in this study. (B) Schematic representation of PCR results for each molecular marker. Polymerase chain reaction products were analyzed using a LabChip GX Touch 24 Nucleic Acid Analyzer after gel purification. White boxes indicate the Saltol QTL region of Mogyang, black boxes indicate the Saltol QTL region of IR64-Saltol and gray boxes indicate the Saltol QTL region of salt-tolerant lines.
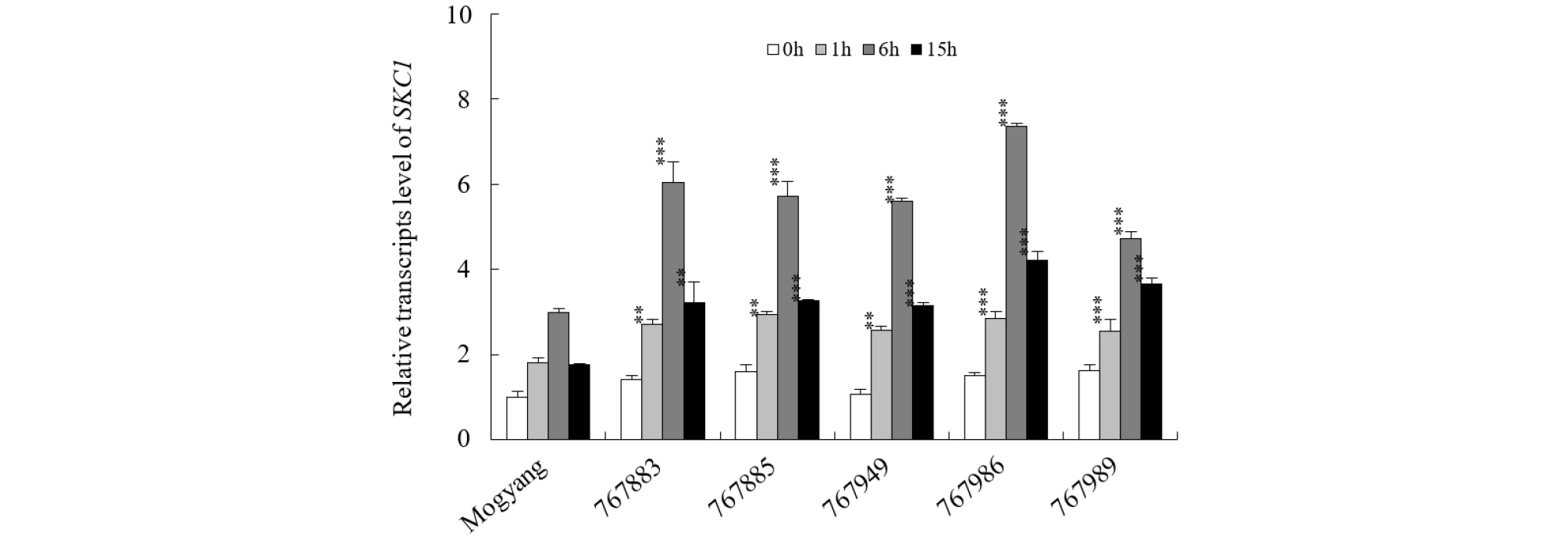
Fig. 2.
Expression analysis of the SKC1 gene from salt-tolerant lines by quantitative real-time PCR. For the salt stress treatment, plants were grown in 0.5x MS medium for 7 days and were treated with 200 mM NaCl for different time periods (Control, 1, 6, and 15 h). Data are means ±SE determined from three independent biological replicates. Asterisks indicate significant differences between Mogyang and salt-tolerant lines at each exposure period determined by Student t-tests (*, p < 0.05; **, p < 0.01; ***, p < 0.001).
Table 3.
Saltol QTL markers on chromosome 1 used in the present study.
Table 4.
Introgression types of Saltol QTL in salt-tolerant lines.
내염성 계통 내 SKC1 발현 양상 분석
벼의 Saltol QTL내 위치한 SKC1은 뿌리의 물관으로부터 유조직 세포로 Na+를 수송하여 Na+/K+ 항상성 유지에 관여하며, 염 스트레스 시 뿌리에서 지상부로 Na+의 이동을 증가시킴으로써 내염성을 증가시킨다(Ren et al., 2005). 내염성 인디카 품종인 Pokkali와 FL478 (Walia et al., 2007), 잡초성벼(Zhang et al., 2018)에서 염 처리 시 야생종에 비해 SKC1 발현이 증가하였다. 이에 근거하여 기내 선발을 통해 확보된 내염성 계통에서 염 스트레스 처리 시 SKC1의 발현 양상을 qRT-PCR을 수행하여 조사하였다(Fig. 2). SKC1의 발현 양상을 조사하기 위해 목양과 선발된 내염성 5계통을 1/2 MS 배지에 치상하여 일주일간 생육 후 200 mM NaCl을 시간별로 처리하였다. 표준 품종 목양의 경우 염 처리 1시간 후부터 SKC1 발현이 증가하여 염 처리 6시간까지 발현이 증가하였고, 6-15시간 사이에 발현이 감소하였다. 무처리구에서는 목양과 내염성 계통 간 발현 차이를 보이지 않은 반면, 염 스트레스 처리 시 내염성 계통 모두에서 목양 대비 발현이 증가하였다. 염 처리 1시간 후부터 선발된 5계통에서 목양 대비 SKC1 발현이 증가하였고, 염 처리 6시간에서 가장 높은 SKC1 발현을 보였다. 또한 염 처리 15시간 후에 SKC1 발현이 감소하지만 모든 계통에서 목양 대비 높은 SKC1 발현을 확인하였다. 이러한 결과로 미루어보아, 선발된 5계통의 내염성은 SKC1 유전자의 발현과 관련이 있을 것으로 판단된다.
적 요
본 연구에서는 간척지와 같은 염류집적 토양에서 재배가 가능한 사료용 벼 품종 개발을 위해 자포니카 우량품종 목양과 내염성 인디카 품종 IR64-Saltol 교배 계통으로부터 기내 선발 방법과 분자마커 분석을 통해 내염성 증진 계통을 선발하였고, 연구결과는 다음과 같다.
1. 목양과 IR64-Saltol 품종에 다양한 농도의 NaCl을 처리하여 신초 길이, 근장 및 생체중의 변화를 분석한 결과, 목양은 IR64-Saltol과 비교하여 50 mM NaCl 농도에서 신초 길이, 근장 및 생체중이 심한 생육 저해를 보였다.
2. 목양과 IR64-Saltol 교배 54계통 224개체를 이용하여 기내 선발 방법을 통해 50 mM NaCl 처리 후 목양 대비 신초 길이, 근장 및 생체중이 양호한 5계통(767883, 767885, 767949, 767986, 767989)을 선발하였다.
3. 내염성 관련 양적형질인 Saltol QTL의 유래를 확인하기 위한 분자마커 분석 결과와 표현형 결과를 비교하여 IR64-Saltol에서 유래된 Saltol QTL이 이입된 계통들은 목양 유래 Saltol QTL이 혼재된 계통들과 비교하여 염 스트레스 시 신초 길이, 근장 및 생체중이 양호함을 확인하였고, 따라서 IR64-Saltol 유래 Saltol QTL의 이입이 내염성을 증진시킨 것으로 판단된다.
4. 내염성 관련 핵심 유전자인 SKC1 발현은 염 처리 후 목양 대비 선발된 5계통 모두에서 높은 발현양을 보이나 목양 유래 QTL이 혼재된 2계통보다 IR64-Saltol QTL만 전이된 3계통에서 보다 높은 발현양을 보였다. 이러한 SKC1의 발현 양상이 선발된 계통들의 내염성에 관여할 것으로 판단된다.
5. 이상의 결과 기내 선발과 분자마커 분석을 통해 선발한 내염성 계통은 간척지와 같은 불량한 환경에서 작물 재배 및 생산이 가능한 내염성 품종 개발의 육종 소재로 활용될 수 있을 것으로 판단된다.




