서 론
재료 및 방법
시험재료 및 재배방법
입형 관련 조사형질
연관지도 작성
QTL 분석
통계분석
결과 및 고찰
육성 집단의 입형 관련 형질 분석
종자 외관 형질 간의 상관관계
입형과 농업 형질들과의 상관분석
연관지도 작성 및 QTL 분석
Haplotype 검정
입형 관련 QTLs의 효과
적 요
서 론
벼는 알곡 상태로 소비되는 대표적인 작물 중 하나로 낱알의 모양은 상품으로서 벼의 외관품질과 매우 깊은 관계가 있다(Yin et al., 2015). 벼 종자의 외관 품질을 결정하는 요인은 종자의 길이, 폭, 두께와 같은 입형과 함께 심복백, 투명도, 완전립 비율 등이 있는데, 이 요인들이 서로 복합적으로 벼의 외관품질 즉 쌀의 상품성을 결정하게 된다(Jiang et al., 2005). 특히 벼 종자 길이에 대한 종자 폭의 비율로 결정되는 종자의 입형은 종자 무게와의 높은 상관으로 인해 수확 후 생산량을 결정하는 주 요인으로 품질과 수량 모두에 관여하는 중요 형질 중 하나로 인식된다(Duan et al., 2014; Gao et al., 2016).
벼의 입형은 벼 생태형에 따라 자포니카 단원형과 인디카 세장형으로 크게 나눌 수 있는데 이러한 차이는 재배지역별 소비자의 쌀 입형 선호도와 일치하는 경향을 보인다(Calingacion et al., 2014). 현재 국내 벼 품종이 길이가 짧고 둥근 단원형 벼가 주를 이루게 된 것은 한국 소비자가 차지고 끈기가 있는 밥을 선호하기 때문이다(Choi, 2002; Kang et al., 2006). 그러므로 소비자의 입형과 취식 선호도는 짧고 둥근 자포니카형을 위주로 벼 품종이 개발이 되게 만들었고 쌀 산업 구조 역시 같은 방향으로 진행되어 왔다고 볼 수 있다(Park et al., 2018). 하지만 입형을 통해 국내 육성 벼 품종을 살펴보면, 고품질을 중심으로 한 국내 벼 품종 육성 전략은 오히려 품종의 입형 및 관련 유전자풀을 단순화하고 좁게 만드는 등의 문제를 낳았다(Park et al., 2017). 현재 쌀 산업은 전세계적인 기후 변화 및 국내 벼 재배환경 변화에 따른 수확기 고온 등숙 문제나 쌀 수출 확대를 위한 전략 다변화가 필요한 실정이다. 이에 따라 다양한 입형 유전자원 확보 등의 현실적 문제가 새로이 떠오른 가운데 국내 벼 품종의 입형 다양화에 대한 요구는 더 커지고 있다.
종자의 입형은 표준화된 기준이 없이 기관이나 국가 마다 다르게 적용되고 있다. 국제미작연구소에서는 현미 장폭비를 기준으로 입형을 4단계(세장형, 중원형, 단원형, 원형)로 구분하고 있으며(IRRI, 2013), 국내는 농촌진흥청의 기준에 따라 5단계(세장형, 장원형, 중원형, 단원형, 원형)로 구분하고 있다(RDA, 2012). 종자 길이에 있어서도 IRRI는 4단계(초장립종, 장립종, 중립종, 단립종), 농촌진흥청은 5단계(초장립종, 장립종, 중립종 중단립종, 단립종)로 각각 다르게 구분하고 있다. 유전적 측면에서는 종자의 입형은 작은 유전력을 가진 다양한 유전자에 의해 복합적으로 조절되는 형질로서 환경변이가 상대적으로 크지 않은 것으로 받아들여지고 있다(Yin et al., 2015). 반면 전통적인 유전 육종에서는 입형이 종자의 길이, 폭, 두께 등에 의해 특징지어 지고 종자의 수량이나 외관품질, 상품성 등과 연계되어 설명되어 왔다(Wan et al., 2005; Fan et al., 2006).
벼 종자 관련 형질은 양적으로 유전되는 것으로 알려져 있는데 지난 수십년간 종자 크기 및 모양을 조절하는 양적유전자좌(QTL)가 400여 개가 탐색되었고 일부는 유전자 수준에서 기능이 규명되었다(Huang et al., 2013; Zhou et al., 2015). 입형과 관련하여 최초로 보고된 유전자는 GS3로서 transmembrane 단백질을 발현함으로서 종자 길이를 증가시키는 것으로 보고 되었다(Fan et al., 2006). DEP1의 경우는 활성 되면 종자의 길이를 감소시키는 쪽으로 작용하는 것으로 알려졌다(Huang et al., 2009). qGL3.1은 이삭의 인산화와 관련하여 종자의 길이 및 무게를 조절한다(Qi et al., 2012).
종자 폭과 관련해서는 2번 염색체에서 GW2가 보고되었다(Song et al., 2007). GW2의 기능 상실로 인해 세포 수가 증가되고 결론적으로 종자 폭 및 중량 증가로 이어진다고 했다. 5번 염색체 상의 qSW5/GW5는 기능 상실 시 종자 폭과 관련하여 sink 크기를 증가시켜 생산량을 증가시킨다고 한다(Shomura et al., 2008; Weng et al., 2008). 이 QTL은 calmodulin 단백질과 상호작용을 가지며 plasma membrane- associated protein을 발현시켜 종자폭을 좁게 만들고 기능을 상실했을 때 폭과 무게가 증가하는 것으로 나타났다. 벼 GS5 역시 5번 염색체에 위치하며 활성 시 종자의 폭 및 중량을 증가시키는 방향으로 관여한다고 하였다(Li et al., 2011). 그 외에도 GW8/OsSPL16 (Wang et al., 2012), GW7 (Che et al., 2015), GLW7 (Si et al., 2016), LGY3 (Liu et al., 2018), GS2 (Che et al., 2015) 등의 유전자가 종자의 크기 및 중량과 관련하여 유전자 수준에서 연구되었다.
벼의 입형은 품질과 수량 두 측면에서 그 역할이 크기 때문에 많은 연구가 계속 수행되었지만 협소한 국내 유전자원의 한계와 시장의 경화된 입형 선호도 등에 의해 현재까지는 논의할 만한 성과가 눈에 띄지 않는 것이 사실이다. 본 연구에서는 국내 벼 품종의 입형을 다양화할 수 있는 새로운 유전자원을 도입 활용하여 입형 관련 특성을 분석함으로써 그 결과를 육종사업에 활용하기 위한 기초 자료를 제공하기 위해 수행되었다.
재료 및 방법
시험재료 및 재배방법
벼 종자 모양 관련 특성을 분석하기 위하여 입형이 세장형인 Langi와 입형이 단원형인 조생자도를 모․부본(Fig. 1)으로 사용하여 mapping 집단을 육성하였다. Langi는 호주에서 도입된 장립 품종으로 밀식 재배에 유리한 수수형이며, 조생자도는 재래도 유전자원 중 하나로서 실험포장에서 계통을 구분하거나 결주를 보충하기 위하여 사용되고 있다. 조생자도는 수술이 영 밖으로 나오지 않은 상태에서 수분 되는 특성이 있어 부본으로 적합하지 않아 Langi를 부본으로 인공교배하여 F1 종자를 생산하였고, 자가수분을 통해 얻은 종자로부터 F2 315개체를 포장에 전개하여 이중 265개체를 유전 분석에 사용하였다.
분석집단은 농촌진흥청 국립식량과학원 벼 시험포장에서 표준 재배법에 준하여 5월 3일에 파종하여 30일간 육묘 후 국립식량과학원 전주 시험포장에 이앙하였다. 시비는 표준시비법(N-P2O5-K2O, 9-4.5-5.7 kg/10a)에 따라 질소는 기비:분얼비:수비를 50:20:30비율로 분시하였고, 인산은 전량 기비로, 칼륨은 기비:수비를 70:30 비율로 분시하였다. 재배관리는 농촌진흥청 표준재배법에 준하여 실시하였다.
입형 관련 조사형질
농업 형질은 F2 plant를 이용하여 조사하였고 출수일에 의한 영향을 배제하기 위하여 Langi와 조생자도 출수일 중간 지점(파종 후 85일)을 기준으로 앞 뒤 5일간 출수하고 임실률이 양호했던 265개체만을 조사 수확하였다. 입형은 조생자도/Langi F2:3세대의 현미를 대상으로 하여 조사하였다. 입형 관련 조사 항목은 입장(길이), 입폭(너비), 입두께(두께) 등을 버니어 캘리퍼스로 10반복씩 실측하였다. 현미 종자 길이가 7.1 mm 이상이면 초장립종, 6.01~7.09 mm이면 장립종, 5.51~6.00 mm이면 중립종, 5.01~5.50 mm이면 중단립종, <5.0 mm이면 단립종으로 구분하였다. 장폭비는 3.0 이상일때 세장형, 2.5~2.99일 때 장원형, 2.0~2.49일 때 중원형, 1.5~1.99일 때 단원형, 1.5미만일 때 원형으로 구분하였다. 종자 무게는 정상립 100개를 각각 3회 반복 측정하여 평균 값을 천립중으로 환산하였다. 주요 농업 특성은 농업과학기술 연구조사 분석 기준에 따라 공시된 F2 개체의 간장, 수장, 수수, 이삭모양 등을 조사하였고 색과 관련하여 엽색, 부선색, 부호영색 등을 조사하였다(RDA, 2012).
연관지도 작성
Langi와 조생자도의 입형 관련 형질을 분석하기 위하여 Kompetitive allele specific PCR (KASP) marker 세트를 이용하여 양친에 대한 다형성 검정을 수행하였다. 연관지도 작성 및 QTL 분석을 수행하기 위하여 양친 검정(parental survey)으로 다형성을 나타낸 marker를 선발하였고, 연관지도는 MapChat 2.32 소프트웨어(Voorrips, 2002)를 사용하여 작성되었다.
QTL 분석
양친사이에서 다형을 나타내는 maker를 이용하여 연관지도를 작성하고 입형 관련 여러 형질들에 대한 표현형을 조사하였다. 조사 결과는 QTL 분석 프로그램인 IciMapping version 4.0 (Meng et al., 2015)을 통해 연관 유전자좌 탐색에 이용되었다. Marker의 그룹핑에는 ICIM의 ‘By LOD’, ‘By Input’ 옵션이 적용되었고, mapping은 Kosambi function으로 수행하였다. 탐색된 QTL의 threshold values는 p>0.05 수준에서 수행된 permutation을 1,000회 test하여 산출된 값을 이용하였고, 각 QTL의 이름은 McCouch가 제안한 명령법에 따라 명명되었다(McCouch, 2008).
통계분석
조사형질들 사이의 관련성을 구명하기 위하여 통계프로그램인 R ver3.6을 이용하여 피어슨 상관 계수를 구하였다. R의 구동 패키지로는 psych가 이용되었으며 3개 이상의 분석을 위해 ‘corr.test’ 명령을 수행하였다. Heatmap 등의 graph작업을 수행하기 위해 ‘corrplot’을 인스톨하였고 Scatterplot을 나타내기 위해 scatterplot3d 패키지가 이용되었다. 평균값의 유의성 검정은 t-test와 Duncan의 다중 평균비교 방법에 의해 p<0.05 수준에서 유의적 차이를 검정하였다.
결과 및 고찰
육성 집단의 입형 관련 형질 분석
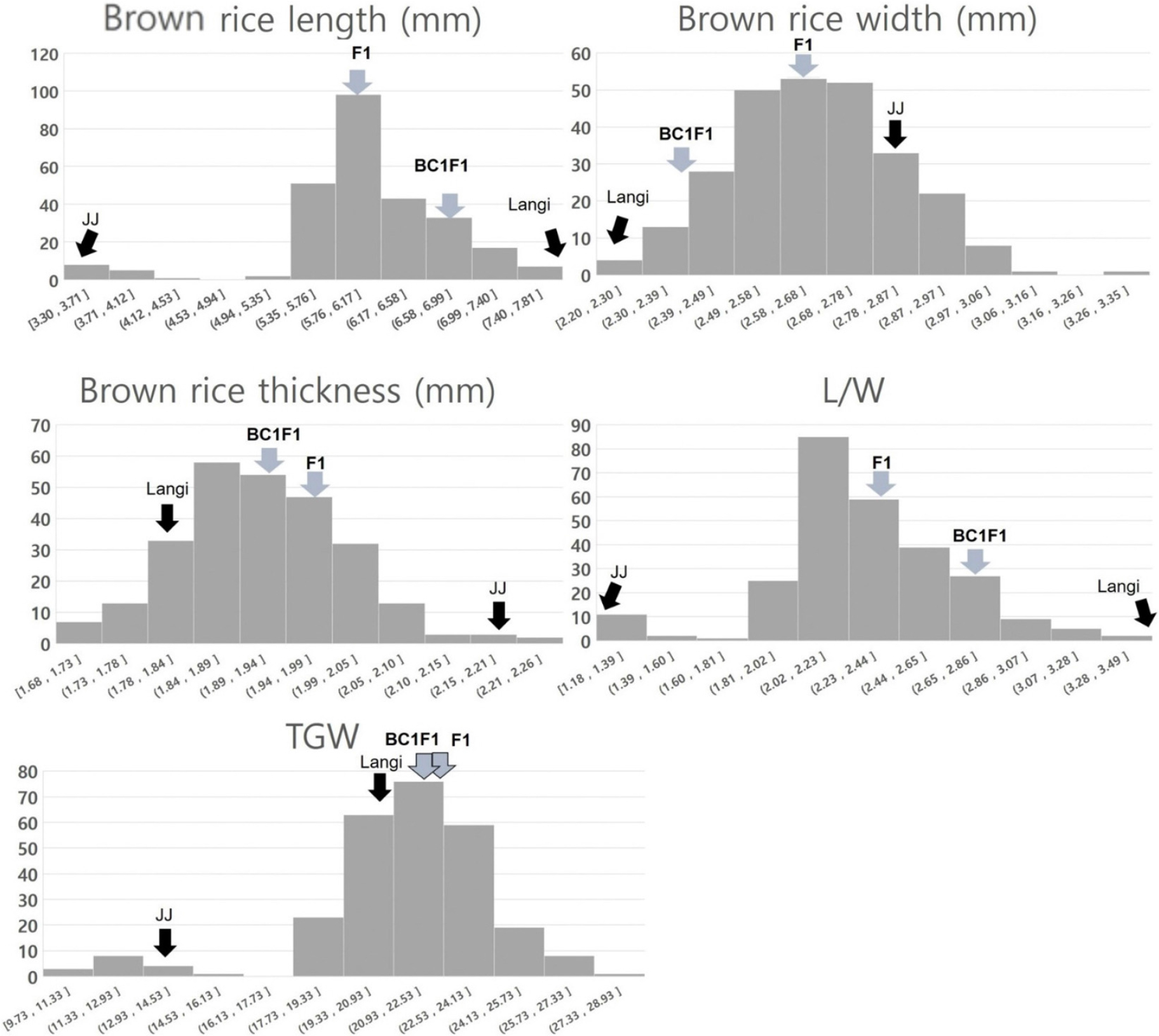
입형과 관련한 주요 형질로 현미의 길이, 너비, 두께, 장폭비, 천립중 등을 조사하였다(Fig. 2). 양친인 조생자도와 Langi는 각각 단립종(2.95 mm)과 초장립종(7.82 mm)이었으며, F2집단의 경우 현미 길이 평균값은 6.04 mm로 3.30~ 7.81 mm 범위에서 정규분포 하였다. F1은 6.11 mm였고 Langi가 여교배된 BC1F1의 경우 6.70 mm로 조사되어 종자 길이가 Langi 쪽으로 수렴하는 것을 확인하였다. 현미의 폭은 2.20~3.35 mm 범위에서 정규분포 하였고 두 교배립의 경우 2.65 mm (F1)와 2.44 mm (BC1F1) 인 것으로 나타났다. 현미의 두께는 1.68~2.26 mm 범위에서 조사되었으며 평균값은 1.92 mm이고, F1과 BC1F1은 각각 1.97과 1.93 mm인 것으로 조사됐다. 장폭비의 경우 1.18~3.49 범위로 원형에서 세장형에 해당되는 것으로 확인되었는데 평균 값은 2.29로 중원형이었고, 두 교배립(F1: 2.35, BC1F1: 2.75)은 중원형과 장원형인 것으로 각각 나타났다.
입형과 관련하여 조사 형질들이 전반적으로 정규분포 양상을 보였으나 초월분리는 크게 나타나지 않았다. 특히 길이 및 장폭비에서는 양친을 초월하는 분리양상이 확인되지 않았는데 이것은 양친간의 차이가 극명할 때 나타나는 현상으로 길이와 관련해서는 양친으로부터 유래한 대립유전자에 의해서만 영향을 받았기 때문인 것으로 보인다. 이것은 단립종과 초장립종인 Nany angzhan/Chuan7 조합 RIL에서 종자 길이와 장폭비 조사에서도 확인되어진다(Bai et al., 2010). 본 연구에서는 길이와 관련하여 원형과 중원형 사이의 단원형(1.5~1.99)입형 비율이 적었다. 이것은 검정 집단내 중단립종 종자의 빈도가 매우 낮았기 때문이다. 두 형질의 빈도 분포표 상에 입장 분포가 bimodal curve처럼 보이고 있는 것을 고려하면 원형에 가까운 조생자도의 입형이 단인자에 의해 독립적으로 작용했을 경우도 생각해 볼 수 있으나 이를 규명하기 위해서는 관련형질에 대한 추가 연구가 필요할 것으로 보인다.
종자 외관 형질 간의 상관관계
현미 천립중과 함께 현미 외관형질들을 조사하여 형질 상호간의 상관관계를 피어슨 법으로 분석하였다(Fig. 3). 분석결과 종자의 길이는 장폭비와 0.89의 강한 정상관을 나타냈고, 종자 폭과는 -0.47의 음의 상관을 보였다. 종자 천립중과는 0.69의 높은 상관을 나타나냈으나 두께와는 상관이 없는 것으로 나타났다. 종자의 폭은 장폭비와 강한 음의 상관(-0.82)을 나타냈고 두께와는 0.43의 중위 정상관을 보였다. 두께의 경우는 다른 형질과 강한 상관을 나타내지는 못하였다. 생산량과 직결된 천립중의 경우 길이(0.69)와 장폭비(0.89)에 정의 상관을 나타낸 반면, 두께 및 폭은 상관이 없었다. 한편, 공시된 육성집단의 입형분포를 확인하기 위하여 종자입형 관현 형질에 대한 산포도를 조사하였다(Fig. 4). 검정 집단에서는 종자의 5가지 입형중 중원형(59%)이 가장 많았으며 다음으로 장원형(24%), 단원형(8%), 원형(5%), 세장형(4%) 순으로 많이 분포하는 것으로 나타났다. 상관분석 결과에서도 알 수 있듯이 입형은 종자길이에 주로 영향을 받아 여러 형태로 분포하고 있었다. 이전 연구에서 입형관련 형질간의 상관관계 결과는 종자길이의 경우 대부분 장폭비와 천립중에서 강한 상관을 나타냈으나 종자폭이나 두께의 경우는 음의 상관(Wu et al., 2018) 혹은 양의 상관 등 육성집단에 따라 다양한 경향이 나타냈다(Bai et al., 2010; Yin et al., 2015). 천립중의 경우 종자 길이나 장폭비 등과는 상관이 높기 때문에 입형 다양화에 있어 종자의 생산량을 증가시키는 측면이 있는데, 본 실험의 결과처럼 길이가 길어질 때 종자의 폭이나 두께가 함께 줄어 든다면 육종계획 수립 시 이들의 상호 작용을 동시에 고려해야 할 것으로 생각된다.
입형과 농업 형질들과의 상관분석
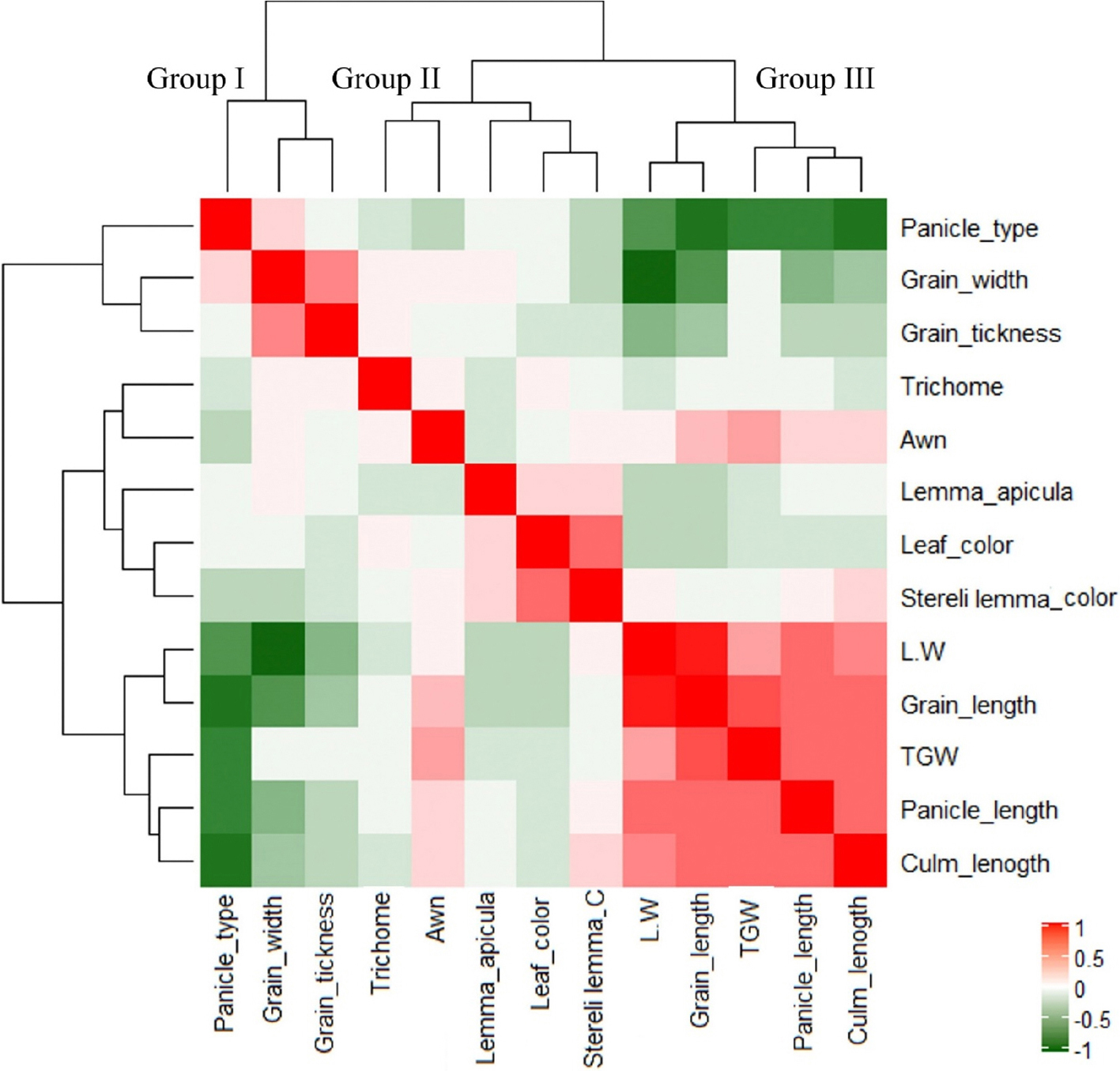
종자 및 벼 외관 관련 총 13개의 형질을 조사하여 각 형질간의 선형관계를 표준화된 공분산분석을 통해 상관분석하였다(Fig. 5). 계층적 군집분석 결과 13개의 형질은 3개의 그룹으로 나눠졌다. 종자의 길이와 장폭비는 천립중 및 간장․수장과 함께 그룹III에 속하며 상호간에 정의 상관을 보였다. 종자의 두께와 폭은 이삭형태와 같은 그룹I으로 묶였고, 모용성, 까락, 엽색, 주두색, 부호영색 등이 그룹II로 분류됐다. 엽색과 주두색은 서로 상관이 높았으며 부호영색과도 정상관을 보였다. 이삭형태, 종자의 너비․두께가 속한 그룹 I은 그룹 III과 음의 상관을 나타냈다.
대체로 조사 형질들은 입형과 서로 독립적으로 작용하는 것으로 보였다. 조사 기관의 발색정도는 안토시아닌 합성기작 조절에 따라 잎, 줄기, 주두, 및 부호영에서 서로 유사하게 발현되면서 기관 상호간에 상관을 가지는 경향이 있는데(Sakamoto et al., 2003; Chin et al., 2016) 본 실험에서도 색 관련 형질들간의 상관관계가 유사하게 나타났다. 한편, 조사한 형질 중 입형과 관련하여 간장과 수장, 이삭모양 및 까락이 입형과 상관을 보였다. 본 연구에서는 조생자도의 단간초형과 짧고 compact한 이삭모양을 가진 개체의 경우 색깔의 유무와 관계없이 종자 길이가 단립이며 입형이 원형에 가까웠는데 이는 heatmap 분석결과와 일치하는 것으로 조생자도에서 식물체 사이즈와 연관된 특정 유전자가 작용함에 따라 식물체 기관에 다면발현된 결과일 것으로 생각된다.
연관지도 작성 및 QTL 분석
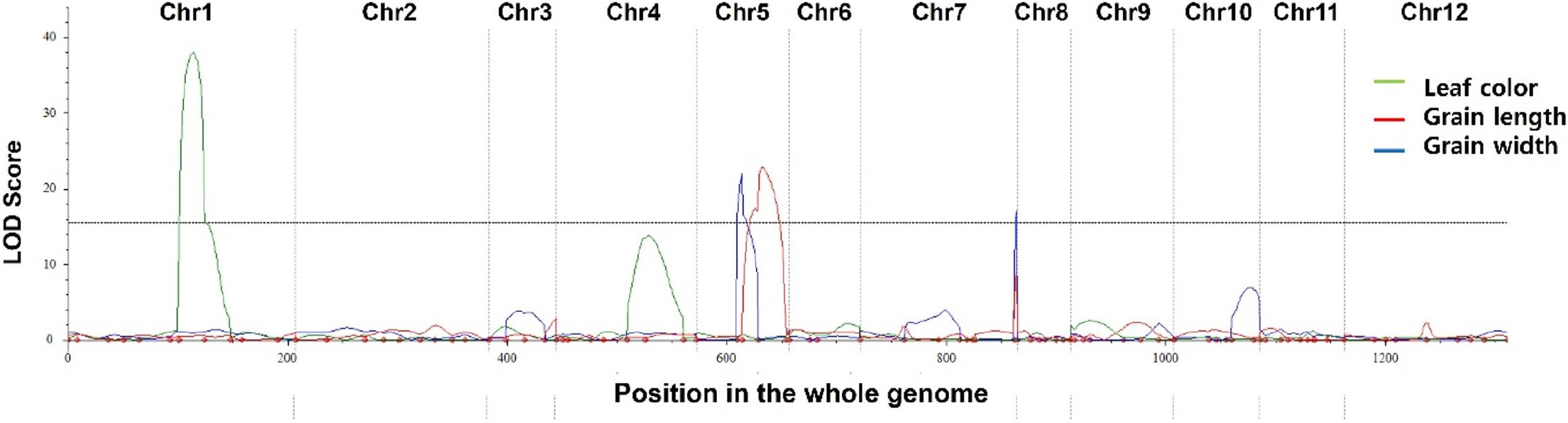
입형 관련 형질의 연관 유전자 좌(QTL)를 동정하기 위하여 조생자도/Langi F2 집단의 연관지도를 작성하였다. 총 771개의 KASP marker를 이용하여 양친간의 다형성을 조사한 결과 215개의 marker가 양친에 다형을 보였다(Table 1). 선발한 marker를 이용하여 육성된 집단에 대한 연관지도를 작성하였고, 이 과정에서 marker의 위치가 중첩되거나 분리비가 편향된 marker들은 제거되어 최종적으로 100개의 marker가 염색체 상에 위치되었다. 염색체별 다형성이 평균 27.9%인 것으로 확인되었고 marker간의 평균 거리는 약 3.3 Mbp였다. 연관지도를 작성 후 조사한 표현형을 함께 분석한 결과 입형과 관련하여 종자의 길이와 폭에서 3개의 QTL이 탐색되었고 엽색과 관련하여 1개의 QTL이 탐색되었다(Table 2, Fig. 6). 종자의 길이 연관 QTL qGL5는 22.78의 LOD 값을 가지고 표현형을 20.31%를 설명하면서 5번 염색체에서 두 marker KJ05_39와 KJ05_40 사이에서 확인되었다. Add 값은 1.36으로 Langi에서 유래한 allele에 영향을 받는 것으로 확인되었다. 종자의 폭과 관련된 두 QTL qGW5와 qGW7은 각각 5번과 7번 염색체에서 동정됐는데 두 QTL은 36.07%와 26.11%에서 각각 표현형을 설명하는 것으로 조사되었다. 엽색과 관련해서는 qLC1이 1번 염색체에서 표현형을 44.93% 설명하며 동정되었다.
Table 1.
Distribution of Kompetitive allele specific PCR (KASP) markers on 12 rice chromosomes exhibiting polymorphisms in the parental survey.
Table 2.
QTLs identified for the traits evaluated in the mapping population.
| Trait | QTL | Marker | LOD | PVE (%)z | Addy | Domx |
| Grain length | qGL5 | KJ05_39 - KJ05_40 | 22.78 | 20.31 | 1.36 | 1.42 |
| Grain width | qGW5 | KJ05_13 - KJ05_16 | 21.93 | 36.07 | -0.18 | -0.01 |
| qGW7 | KJ07_73 - KJ76_76 | 16.70 | 26.11 | -0.14 | -0.01 | |
| Leaf color | qLC1 | KJ01_73 - KJ01_83 | 55.67 | 44.93 | -3.73 | -3.55 |
본 연구의 입형 QTL은 5번과 7번 염색체에서 탐색되었는데, 이전 연구에서는 qSW5/GES5 (Shomura et al., 2008; Duan et al., 2017)와 GS5 (Li et al., 2011)가 5번 염색체에서 보고되어 종자의 폭과 무게에 영향을 주는 것으로 확인 되었고, 7번 염색체에서는 GW7/GL7 (Wang et al., 2015)과 GLW7/OsSPL13 (Si et al., 2016)이 종자 크기 증가와 관련하여 보고된 적이 있다. 염색체 상 위치로 비교해 보면 5번 염색체 short arm에 탐색된 qGW5는 기존의 qSW5/GES5이나 GS5와 물리적 거리가 2 Mbp 떨어져 있으며 5번 염색체 long arm에 유치한 qGL5는 기존에는 보고된 적이 없는 새로운 입형 QTL인 것으로 보인다. 다만 Aya et al. (2014)이 N-methyl-Nitrosourea (MNU)에 의한 새로운 loss-of-function 돌연변이체를 보고하면서 auxin signaling pathway에 관여하는 small organ size1 (smos1) 유전자가 cell extension에 관여한다고 하였는데 qGL5의 flanking region이 smos1을 포함하고 있다. SMOS1 대립유전자인 smos1은 돌연변이로 정상 기능이 상실되어 세포의 size를 작게 함으로써 벼의 초장을 포함하여 여러 기관의 크기를 감소시키는 역할을 한다고 알려져 있다. 본 연구에서는 smos1에 관한 실험을 수행하지는 않아 상호간의 연관성을 입증하지 못하였지만 조생자도의 극단적 초형 및 입형을 고려할 때 qGL5와 smos1과의 연관성을 추정해볼 수 있으나 qGL5가 단독이든 복합적인 방식이든 Langi/조생자도 집단의 입형 변이에 관여했을 것으로 생각된다. 엽색과 관련하여 동정된 qLC1은 기존에 알려진 유전자(Hadagal et al., 1981; Sakamoto et al., 2003)와 위치가 동일한 것으로 확인되었다.
Haplotype 검정
종자 입형에 영향을 주는 것으로 알려진 8개 유전자의 분자표지를 이용하여 Langi와 조생자도에 대한 대립유전자형 패턴을 분석하였다(Table 3). 장립종인 Langi의 경우 qSW5를 제외하고 신동진과 동일한 대립유전자 형을 나타냈다. Shomura et al. (2008)는 qSW5가 대부분의 온대 자포니카 생태형 벼에서 특정 염기서열이 결실(1,212 bp)된 형태(qsw5N)로 존재하며 이때 종실의 무게와 폭을 증가시키는 방향으로 입형에 관여한다고 하였는데, 단원형이 대부분인 국내 자포니카 벼 품종(236품종)은 약 98.7%가 qsw5N 형으로 qSW 대립유전자형은 자포니카 생태형에서는 매우 드문 유전자형이다(Lee et al., 2020). 따라서 자포니카 생태형이며 장립종인 Langi는 동정된 QTL, qGL5외에 qSW5 (Shomura et al., 2008)가 작용하여 초장립 세장형 입형을 가지게 된 것으로 추정할 수 있다. 이 것은 qSW5부근에서 탐색된 qGW5가 Langi가 아닌 조생자도에서 유래하여 종자의 폭에 영향을 주었다는 본 연구 결과와도 일치하는 것이다. 이 결과는 국내 벼 품종의 입형 다양화를 위한 육종사업에 중요한 정보를 제공할 것으로 생각된다. 한편 조생자도의 경우는 8개의 유전자가 일품과 동일한 대립유전자 형인 것으로 나타났다. 일품의 유전자 조합은 단원형이 대부분인 국내 벼 품종 중 가장 일반적인 조합(47%)으로(data not shown), GW7을 제외할 경우 국내 236개 자포니카 품종의 90%가 가지고 있는 입형 유전자 조합이다(Lee et al., unpublished data). 따라서 haplotype 결과가 조생자도의 입형을 설명하지 못하는 것은 새로운 유전자의 이입보다는 smos1 (Aya et al., 2014)과 같은 특정 유전자의 변이가 입형에 관여한 결과임을 가정해 볼 수 있다. 표현형적으로 일반적인 자도의 입형이 단원형인데 반해 조생자도의 입형만 원형인 점이나 유전분석에서 종자 길이나 3차원 산포도 분석 결과가 bimodal curve 형태로 입형이 원형인 계통만 구분한다는 것도 근거가 될 것으로 생각된다.
Table 3.
Haplotype testing of japonica cultivars including parents according to allele types of eight genes associated with grain shape.
| Gene | Target traits | Allele type (effect)z | Allele type of plants | Reference | |||
| Langi | Josaeng-jado | Shindongjin | Ilpum | ||||
| GW2 | Grain- | gw2WY3(+) | (Song et al., 2007) | ||||
| Width & Yield | GW2FAZ1(N) | GW2 | GW2 | GW2 | GW2 | ||
| GS3 | Grain- | gs3A-allele(+) | gs3A | gs3A | (Fan et al., 2006) | ||
| Length & | GS3B-allele(M) | ||||||
| Weight | GS3C-allele(N) | GS3C | GS3C | ||||
| qGL3 | Grain- | qgl3N411(+) | (Qi et al., 2012) | ||||
| Length | qGL39311(N) | qGL3 | qGL3 | qGL3 | qGL3 | ||
| qSW5 | Grain- | qsw5_NNipponbare(+) | qsw5_N | qsw5_N | qsw5_N | (Shomura et al., 2008) | |
| Width & | qsw5_IIndicaⅡ(M) | ||||||
| Thickness | qSW5Kasalath(N) | qSW5 | |||||
| GS5 | Grain- | gs5H94(N) | gs5 | gs5 | gs5 | gs5 | (Li et al., 2011) |
| Length &L/W | GS5Zhensan97(+) | ||||||
| TGW6 | Grain- | tgw6Kasalath(+) | (Ishimaru et al., 2013) | ||||
| Weight | TGW6Nipponbare(N) | TGW6 | TGW6 | TGW6 | TGW6 | ||
| GW7 | Grain-Width | gw7Nipponbare(+) | gw7 | gw7 | (Wang et al., 2015) | ||
| & Thickness | GW79311(N) | GW7 | GW7 | ||||
| GW8 | Grain-Width | gw89311(N) | (Wang et al., 2012) | ||||
| & Yield | GW8Nipponbare(+) | GW8 | GW8 | GW8 | GW8 | ||
입형 관련 QTLs의 효과
각 QTL의 식물체 이입 여부에 따른 QTL의 효과를 검정하기 위하여 목표 형질의 특성 변화를 통계적으로 분석하였다(Fig. 7). 종자의 길이와 관련하여 qGL5가 포함된 그룹은 그렇지 않은 그룹에 비해 길이 신장효과가 유의하게 증가하였다(Fig. 7a). 장폭비에 있어서는 qGW5와 qGW7이 모두 있는 그룹과 qGW5가 단독으로 있는 그룹이 QTL 모두 없거나 qGW7만 단독으로 있는 그룹에 비해 종자 폭 신장 효과가 있는 것으로 확인되었다(Fig. 7b). 장폭비의 경우 종자 폭 증가로 인해 두 QTL 그룹(qGW5+qGW7, qGW5)에서 다른 그룹에 비해 장폭비 값이 유의하게 감소하는 경향을 보였다. 종자 길이와 폭 신장은 장폭비, 즉 입형 결정에 있어 서로 음의 상관이 있었기 때문에 상호간의 영향을 주며 작용하는 것을 확인하였다(Table 4). QTL이입 여부에 따른 입형은 단원형(1.5~1.99), 장원형(2.0~2.49), 세장형(>3.0)으로 분포하였는데, qGL5 (+) 경우는 qGW 존재 여부와 관계없이 종자 길이 신장에 영향을 주는 것으로 나타났다. 한편, 두 QTL (qGW5;qGW7)의 조합을 고려할 경우 장원형 세장형 등으로 구분이 가능하였는데 교배 조합이 다를 경우 더 다양한 입형도 기대해 볼 수 있다. 단원형이 주류를 이루고 있는 국내 벼 품종의 입형 다양화를 위해 자포니카 형인 Langi를 포함하는 조합이 구성된다면 본 연구 결과를 활용하여 국내 벼 품종 입형 유전자형 확대 및 입형 다양화에 기여할 것으로 전망한다.
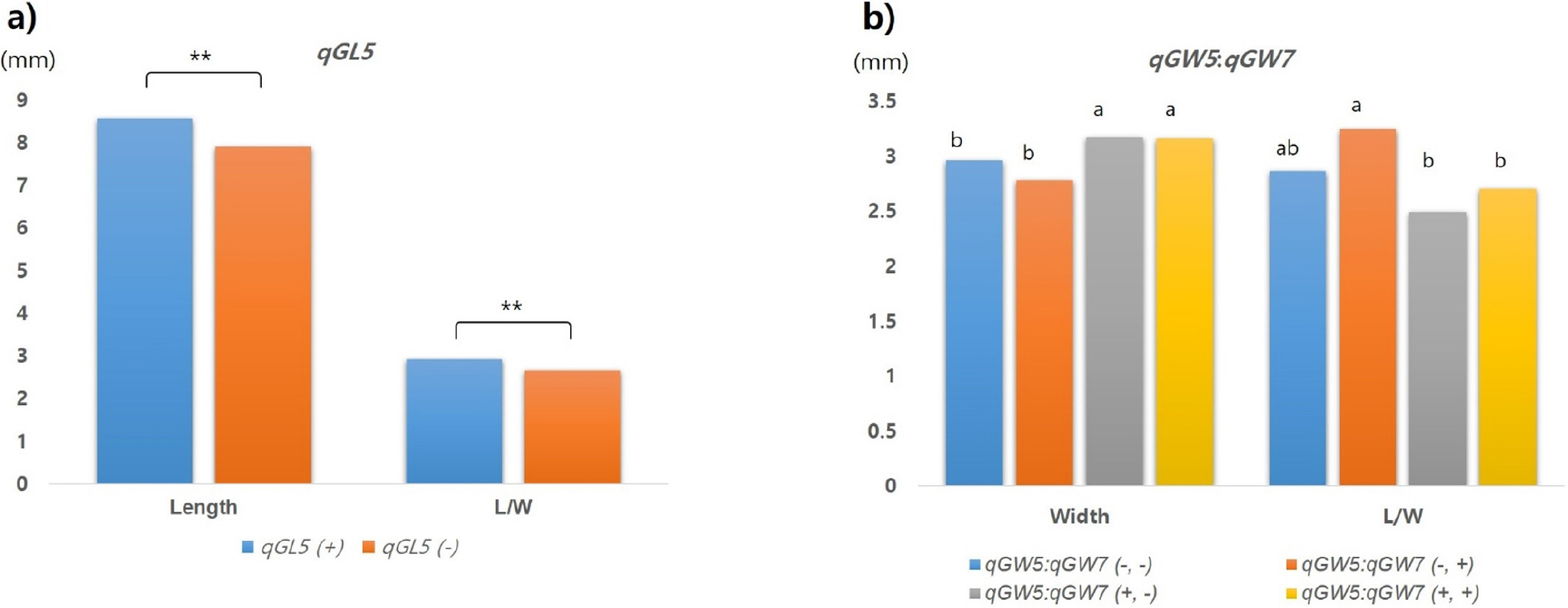
Fig. 7
QTL effect for grain length (a) and width (b) by the presence of the detected QTL. qGL5 (+) was derived from Langi homozygous allele which increases grain length. The QTLs, qGW5 (+), and qGW7 (+), were derived from Josaeng-jado homozygous alleles which increase grain width. ** mean significantly different at p<0.05 by t-test. The letters from a to b indicate significantly different values according to statistical analysis using Duncan’s Multiple Range Test (alpha = 0.01) in R.
적 요
본 연구는 단조로운 국내 벼 품종의 입형을 다양화하기 위하여 초장립종 자포니카 품종을 활용해 그 특성을 국내 벼 품종에 도입함으로써 입형이 다양한 새 품종을 개발하려는 기초 육종연구의 일환으로 수행되었다. 입형 다양화를 위하여 세장형 자포니카 벼 품종인 Langi와 입형이 극단적으로 원형에 가까운 자포니카형 조생자도를 교배하여 유전분석 및 QTL 탐색을 위한 mapping 집단을 육성하였고, 이를 이용하여 입형 관련 형질의 상관분석, QTL 분석, validation test 및 탐색된 QTL 효과를 검정한 결과는 다음과 같다.
1. 조생자도와 Langi 조합의 교배립을 생산하여 F2 집단을 육성하였고 출수 일수를 고려하여 최종 265 개체를 선발․수확하여 주요 형질에 대한 조사와 QTL 분석에 이용하였다.
2. 육성된 mapping집단의 입형 관련 형질들은 종자 길이에 대해 종자 장폭비(0.89)와 천립중(0.69)이 상대적으로 높은 상관계수를 나타냈고, 종자폭에 대해서는 종자 길이(-0.47)와 장폭비(-0.82)가 음의 상관을 나타냈으며 종자 두께(0.43)는 정상관을 보였다.
3. QTL 분석 결과 종자 길이와 관련하여 벼 5번 염색체에서 PVE 값이 20.31%인 qGL5와 7번 염색체에서 PVE 값이 각각 36.07%와 26.11%인 qGW5와 qGW7을 탐색하였다. qGL5는 세장형인 Langi로부터 유래하였으며, 두 QTL (qGW5, qGW7)은 원형인 조생자도로부터 유래하였다.
4. Validation test결과 Langi는 자포니카형 벼(qsw5_N)에서는 매우 드문 qSW5형 대립유전자를 가지고 있으며 이것은 qGL5와 함께 작용하여 집단내에서 종자 길이신장에 관여했을 것으로 추정된다.
5. 탐색 QTL의 효과는 qGL5 (+)와 qGW5:qGW7 조합에서 중원형, 장원형, 세장형 등 입형의 변이가 다양하게 나타났는데 Langi가 자포니카 생태형이란 점과 QTL의 효과가 증명되어 MAS활용이 가능하다는 점 등 입형 다양화를 위한 육종사업에서의 효과 및 활용도가 다양할 것으로 생각된다.