서 론
재료 및 방법
수발아율 검정
RNA-Sequencing 시료
Total RNA 추출
DEGs 선발 및 기능 분석
Quantitation Real-Time PCR 분석
ABA 함량 분석
결과 및 고찰
적 요
서 론
벼의 수발아는 등숙 기간 중에서 황숙기와 완숙기 사이에 발생하기 쉬우며(Park & Park, 1984), 우리나라와 같은 온대지역에서는 벼 등숙기에 발생하는 태풍이나, 고온과 잦은 강우를 동반하는 이상기상으로 인해 수발아 피해가 발생한다고 한다(Suh & Kim, 1994; Park & Kim, 2009). Ju et al. (2000)은 출수 후 45일에 25/15°C의 변온을 갖는 항온항습기에서 이삭을 6일 동안 치상하여 조사하는 실내검정방법으로 포장검정과 고도의 상관 관계를 보이므로 효율적인 검정방법이라고 하였다.(품종의 조만성에 따라서는 조생종이 중만생종보다 수발아성이 크다는 보고도 있으며(Park & Park, 1984), 조생종에도 수발아성이 낮은 품종이 있다는 보고(Park & Kim, 2009)도 있어 생태형별로 정확하게 구분이 되지 않는다. 벼 수발아는 등숙환경의 영향을 크게 받지만 기상조건에 따른 수발아성 변이에 관한 보고가 없으며, 현행 수발아 검정기준(RDA, 2012)은 출수 후 40일로 생태형에 관계없이 동일하므로 출수기가 다른 품종 간에 종자의 생리적인 등숙상태가 일정하지 않은 단점이 있다(Kang et al., 2018). 수확 전 이삭이 달려있는 상태에서 도복이나 침수에 의한 종자의 발아는 쌀의 품질과 생산성의 악영향을 끼친다고 한다(Kim et al., 2008). 수발아는 품종이나 등숙기간에 차이를 보이나 수확전인 출수 후 55일 이후에 높은 수발아율을 보인다(Baek & Chung, 2014). 수발아율이 높을수록 수확 후 발아율과 출아율, 종자활력 등이 감소하며 변색이 진행 되며 미발아율과 부패율이 높아지고, 한번 수발아된 종자는 발아율 및 입모율이 크게 떨어지므로 종자로 사용하기 어려워진다(Shon et al., 2014). 벼 수발아가 발생하면 완전립률과 쌀 외관품질이 저하되며 현미천립중이 감소하여 수량도 저하된다고 한다(Lee et al., 1985, Oh et al., 1987, Kim et al., 2008). 수발아 유아가 3 mm까지 발생할 때 파종기 발아율과 출아율의 감소가 크고, 수발아 유아가 3 mm 이상에서 더욱 크게 발아율과 출아율이 감소될 것으로 보고하였다(Shon et al., 2014).
종자의 휴면성은 발아가 유리한 환경조건 하에서도 발아를 하지 않는 적응형질이다. 휴면 상태가 낮은 비휴면종자는 최적의 환경조건하에서 쉽게 발아하며 이러한 특성은 작물의 수확량과 품질을 크게 감소시킨다. 이러한 종자의 휴면 유도, 유지 및 타파는 광범위한 내적 환경적 요인을 받는 복잡한 생리학적 과정이며 식물 호르몬인 abscisic acid (ABA)와 gibberellin (GA)는 종자 휴면과 발아의 조절에 길항적으로 작용하는 주요 내인성 인자이다(Kucera et al., 2005). 수발아를 일으키는 기전연구에서 선발된 돌연변이체는 ABA의 카로티노이드 전구체의 생합성에 필수적 효소들로 각각 Phytoene desaturase (OsPDS), zeta-carotene desaturase (OsZDS), carotenoid isomerase (OsCRTISO), lycopene beta-OsLCY (beta-OsLCY)를 코딩하는 유전자가 망가졌으며, 4개의 돌연변이체 모두에서 ABA의 양이 야생형에 비해 감소된다고 한다(Fang et al., 2008). 보리의 수발아 연구에서 ABA의 이화단계를 촉매하는 HvCYP707A1은 침윤에 반응하여 발현이 증가 하여 ABA의 감소를 일으켜 높은 발아율을 보인다고 한다(Chono et al., 2006). ABA와 GA 사이의 상호 관계에 대한 연구에서 애기장대의 Abscisic acid-insensitive-4 (ABI4) 돌연변이(abi4)는 야생형의 비해 훨씬 빨리 발아하며 abi4의 자엽은 야생형보다 빠르게 발달한다. abi4는 GA 생합성 억제제인 paclobutrazol (PAC)에 대한 내성을 보이며 ABI4 과발현 돌연변이체는 PAC에 민감하다. 외부적으로 처리한 GA는 ABI 과발현체의 발아를 촉진 시킨다(Shu et al., 2013). 수발아율과 α-amlylase 활성은 상관관계가 있으며 수발아성이 높은 품종에서 α-amlylase 활성이 더 높았다(Ko et al., 2005). α-amylase 활성을 억제한다고 알려진 eugenol 처리 실험에서 eugenol를 처리할수록 α-amylase 활성이 억제 되고, 발아속도가 현저히 줄어들었다(Hu et al., 2017). 국내 품종의 벼 수발아연구는 품종간 수발아성 변이 연구가 활발히 이루어 졌으나 유적학적, 생리학적 연구가 미흡한 실정이다. 따라서 본 연구는 수발아성이 높은 조평과 내수발아성이 강한 조운을 대상으로 출수 후 적산온도와 검정온도에 따른 수발아 발생 양상 변이를 조사하고 RNA- Sequencing방법을 이용하여 유전학적, 생리학적으로 구명하고자 한다.
재료 및 방법
수발아율 검정
시험품종으로는 조평과 조운을 사용하였다. 정밀유리온실에서 동일한 조건(29/19°C)으로 출수 후 수발아 처리 지점까지 생육하였다. 수발아 처리는 이삭별로 출수일을 기록한 이후 출수일이 동일한 이삭으로 실험에 사용 하였으며, 검정방법은 플라스틱 상자 안에 탈지면 시트를 2겹으로 깔고 그 위에 이삭을 올려 놓고 다시 탈지면 시트를 덮었다. 탈지면 시트가 마르지 않도록 물을 공급하여 포화 습도를 유지 하였다. 수발아 처리 시기는 출수 후 등숙기간 따른 수발저항성을 알아보기 위해 등숙시 적산온도 700, 900, 1100°C에 처리하였다. 검정온도는 변온인 25/15°C, 27/17°C, 29/19°C로 다르게 하고 처리 후 4일부터 10일까지 3일 간격으로 같은 시간대에 조사하였다.
RNA-Sequencing 시료
분석용 시료는 수발아 검정시료와 동일하게 재배한 출수 후 등숙기간이 적산온도 900°C인 조평을 사용하였다. 수발아 검정실험과 동일한 조건하에서 3일간 27/17°C에서 포화습도를 유지한 후 종자만 채취 하였다. 대조군으로 수발아 처리가 되지 않은 종자를 사용하였다. Total RNA를 추출 후 RNA-Sequencing을 진행하였다(Seeders, Daejeon).
Total RNA 추출
0.1 g의 종자를 액체 질소로 얼려 막자 사발에 분쇄하여 Plant RNA extraction kit (Sigma)를 사용하여 제조사에서 제공한 방법에 따라 추출하였다. Turbo Dnase (Invitrogen)을 사용하여 chromosomal DNA를 제거 후 RNA clean-up kit (MACHEREY-NAGEL)로 제조사에서 제공한 방법에 따라 사용하여 정제 하였다.
Sequencing pre-processing, Read mapping, Normalization, Annotation
시퀀싱 된 transcriptome short reads는 cutadapt로 adaptor 서열을 제거하고, SolexaQA package의 DynamicTrim과 LengthSort를 이용하여 전처리를 수행하였다. 전처리 과정을 통과한 cleaned reads를 mapping하여 유전자가 발현되는 정도를 나타내는 발현값(read count)을 계산하였다. Mapping은 HISAT2 software를 사용하였으며, HTSeq (v.0.11.0)를 사용하여 각 유전자에 mapping된 reads의 총수로 발현값을 측정하였다. 데이터편차가 존재하는 샘플의 적절한 유전자 발현값을 계산하기 위해 총 개수에 대한 normalization을 수행하였다. Normalization 방식은 R의 DESeq library을 사용하였고, 유전자의 기능을 파악하기 위해 RAP-DB (https://rapdb.dna.affrc.go. jp/)에서 제공하는 annotation 정보를 이용하였다.
DEGs 선발 및 기능 분석
샘플 간 유의하게 발현하는 유전자(DEGs; Differentially Expressed Genes, Anders & Huber, 2010)선발은 각 유전자에 mapping된 발현값이 상호간에 비교되는 샘플에서 2배 이상 발현의 차이를 확인하는 2 fold change 방법과, adjust P-value (FDR)이 0.01 이하를 만족하는 binomial test 방법을 동시에 사용하였다. 선발된 DEGs의 Gene ontology (Ashburner et al., 2000) 분석을 위해 Reference에서 제공하는 Gene ontology 정보를 이용하였으며 in house scripts를 통해 Gene ontology 분석을 수행하였다. Significance level은 0.05로 지정하여 functional category인 BP (Biological Process), CC (Cellular Component), MF (Molecular Function)로 분류하였다. 선별된 DEG 정보를 바탕으로 KEGG (Kyoto Encyclopedia of Genes and Genomes) mapper를 이용하여 특정 경로의 유전자 발현 패턴을 확인하였다.
Quantitation Real-Time PCR 분석
추출한 1 µg의 RNA를 iScript™ cDNA Synthesis Kit (Bio-Rad)를 사용하여 제조사가 제공한 방법에 따라 cDNA를 합성하였다. 총 20 µl의 반응액을 PCR기계를 이용하여 역전사 시킨 후 nuclease-free water를 180 µl 첨가하여 1/10으로 희석하였다. mRNA의 정도를 알아보기 위하여 합성된 cDNA중 5 µl를 주형으로 삼고, 10 pmole의 상보적 primer (Table 1)를 0.5 µl와 SYBR®Green Realtime PCR Master Mix (Toyobo)를 혼합하여 Quantitation Real-Time PCR을 수행하였다. 각 유전자의 Ct값을 reference 유전자인 ubiqutin의 Ct값으로 보정한 뒤 비교하였다.
Table 1.
Primers used in this study.
ABA 함량 분석
마쇄한 종자 분말 0.05 g에 80% 메탄올 1 ml을 첨가하여 4°C에서 교반하면서 약 16시간 동안 추출하였다. 추출액을 5,000 g로 5분간 원심분리하여 얻은 상청액을 취하여 메탄올과 증류수가 제거될 때까지 건조시켰다. 건조된 시료는 Tris Buffered Saline (TBS) 1 ml에 재현탁시켜 ABA 측정용 시료로 사용하였다. ABA 함량은 Phytodetek ABA Test Kit (Agdia)를 사용하여 제조사가 제공한 방법에 따라 추출한 후 405 nm에서 측정된 흡광도를 측정하였다(Walker- Simmons, 1987).
결과 및 고찰
조운과 조평의 출수후 등숙시 적산온도 및 검정온도에 따른 수발아율은 Table 1과 같이 출수후 등숙시 적산온도가 축적될수록, 검정온도가 높을수록 수발아 저항성이 낮아졌다. 수발아 발생시 초반 대응이 중요하기 때문에 4일째 수발아 비율을 품종의 수발아 저항성을 나타내는 지표로 정하였다. 처리후 4일째 등숙시 적산온도별로 살펴보면 적산온도 700°C 실험에서 25/15°C 처리시 조운은 0.2% 매우 낮은 수발아율을 보이고, 조평도 1.3%의 낮은 수발아율을 보인다. 27/17°C 처리시 조운은 0.8%, 조평은 5.7%의 수발아율을 보이며, 29/19°C 처리시에는 조운은 1.3%, 조평은 16%의 수발아율을 보인다. 적산온도 700°C 에서는 비교적 두 품종 모두 수발율이 낮았지만 조운보다 조평이 수발아율이 높고 검정온도가 높아질수록 수발아 발생 비율이 높았다. 등숙시 적산온도 900°C에서 25/15°C 처리시 조운은4.8%의 낮은 수발아율을 보이는 반면 조평은 30.7% 상대적으로 높은 수발아율을 보인다. 27/17°C 처리시 조운은 9.5% 수발아율을 보이며 조평은 58.1%로 절반이 넘는 종자가 수발아 하였다. 29/19°C 처리시 에서도 조운은 12.8% 수발아율을 보인 반면 조평은 67.2%의 높은 수발아율을 보였다. 등숙시 적산온도 700°C 처리시와 같은 경향성을 보였으며, 등숙시간이 길어질수록 수발아 발생율이 높아졌다. 등숙 후반부인 적산온도 1100°C에서도 25/15°C 처리시 조운은 5%의 수발아율을 보인 반면 조평은 59.6%로 상대적으로 낮은 온도에서도 수발아율이 높았다. 27/17°C, 29/19°C 처리시에서도 조운은 20.8%, 42.4%로 수발아율이 상승은 하였지만 조평은 60.9%, 87.1%로 수발아율이 상승하였다(Table 2). 두 품종 모두 출수후 등숙시 적산온도 축적될수록, 검정온도가 높을수록 수발아율이 상승하였지만 조평이 상대적으로 모든 조건에서 조운보다 수발아율이 높았다.
Table 2.
Pre-harvest sprouting (PHS) rate in rice cultivars according to the Winkler scale after heading and test temperature.
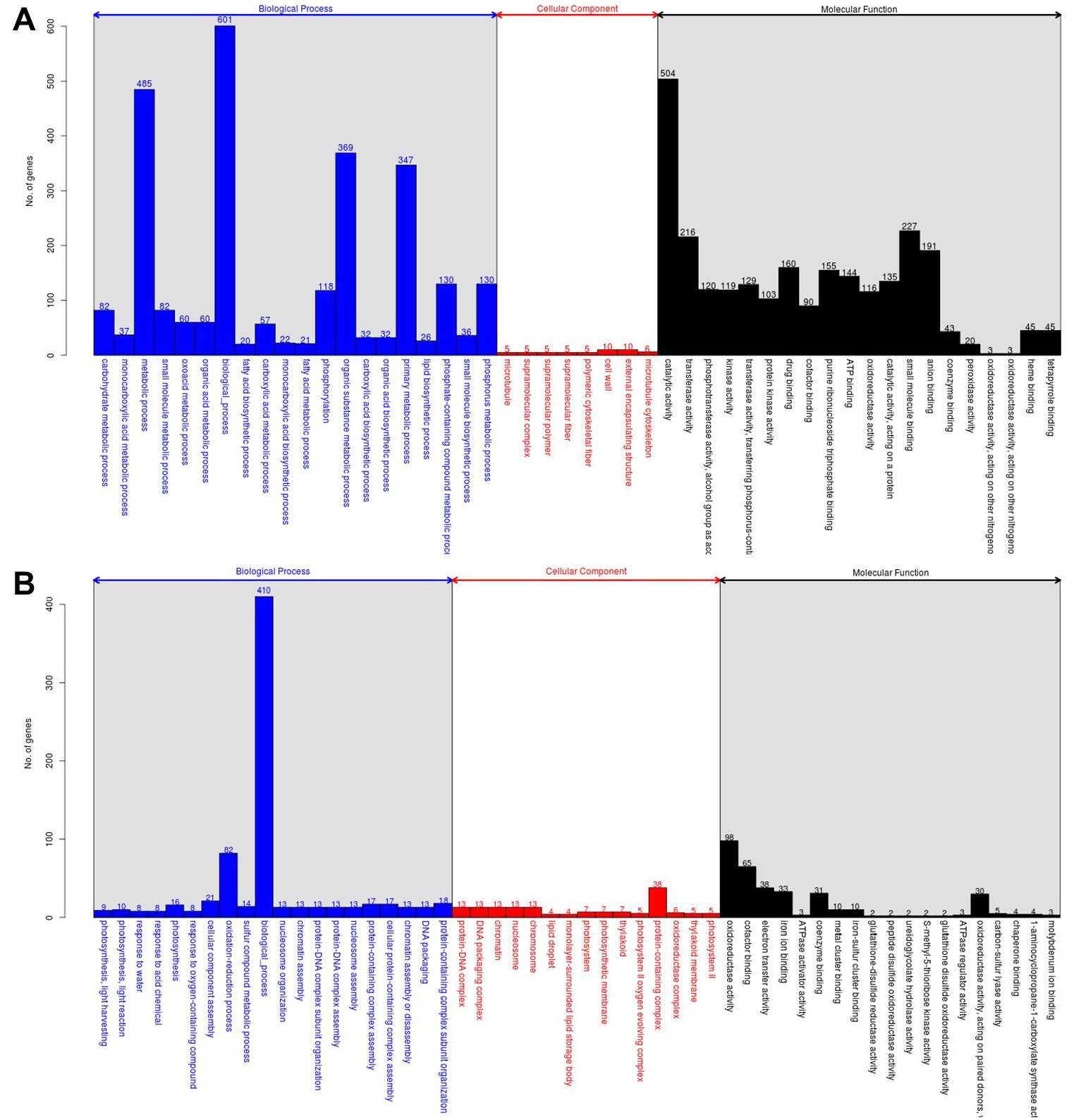
수발아 저항성이 낮은 조평이 수발아가 나타날 때 전체 유전자의 발현을 살펴보기 위해 RNA sequencing을 진행하였다. Trimming된 short read의 수는 16,693,329에서 24,499,743개, 전체 길이는 2,029,653,355 bp에서 3,338,876,276 bp로 평균 129.86 bp에서 136.36 bp였다. 전체 raw data에서 trimming하여 남은 raw data 비율은 82.11%에서 86.66%로 25 bp 이하의 read 제거하였다(Table 3). 표준 유전자 세트로 사용한 벼 유전자 서열은 RAP (https://rapdb.dna.affrc.go.jp/)에서 제공하는 database (IRGSP -1.0_genome)를 사용하였다. 분석에 사용한 표준 유전자 37,657개 중 발현값을 갖는 유전자는 32,388개이며, 모두 기능을 예측할 수 있는 유전자였다. 표준 유전자 세트에 trimmed read를 mapping하여 발현값을 계산하였으며 평균적인 mapping 비율은 95.46%였다(Table 4). 수발아된 종자에서 발현차이가 유의하게 나타나는 유전자군(DEGs: Differentially Expressed Genes)을 살펴보면 up-regulation되는 유전자는 1,581개로 나타났으며, down- regulation되는 유전자는 1084개로 모두 기능이 알려진 유전자들이었다(Table 5). 선별된 DEGs의 전체적인 기능을 알기 위해 Gene Ontology (GO)에서 해당 DEGs를 biological process와 cellular component 그리고 molecular function으로 분류하였다. 수발아 시 up-regulation된 DEGs의 biological process분류에 따르면 특정되지 않은 biological process에 포함되는 유전자가 601개로 가장 많았고, metabolic process가 485개, organic substance metabolic process 369개 순으로 많았다. cellular component 분류에서는 Biological process에 비해 해당되는 분류의 유전자가 5~10개로 상대적으로 적었다. Molecular function 분류에서는 catalytic activity에 관여하는 유전자가 504개로 가장 많았고 small molecule binding 유전자 227개, transferase activity에 관여하는 유전자가 216개 순으로 많았다(Fig. 1A). 반대로 수발아시 down- regulation된 DEGs의 Biological process분류에는 특정되지 않은 Biological process에 포함되는 유전자가 410개로 가장 많았고, oxidation reduction process에 관여하는 유전자가 82개로 많았다. Cellular component 분류에서는 protein- containing complex에 속하는 유전자가 38개 가장 많았다. Molecular function 분류에서는 oxidoreductase activity에 관여하는 유전자가 98개로 가장 많았고, cofactor binding에 속하는 유전자가 65개, electron transfer activity 관련 유전자가 38개 순으로 많았다(Fig. 1B).
Table 3.
Trimmed sequence data.
| Sample | Description | Number of reads | Total length (bp) | Average length (bp) | *Trimmed/raw (%) |
| Jopyeong | Control 1 | 16,693,329 | 2,275,282,352 | 136.30 | 86.66 |
| 16,693,329 | 2,181,451,989 | 130.68 | 83.08 | ||
| Control 2 | 24,499,743 | 3,338,876,276 | 136.28 | 85.17 | |
| 24,499,743 | 3,181,517,610 | 129.86 | 82.11 | ||
| PHS 1 | 17,587,728 | 2,389,827,494 | 135.88 | 86.49 | |
| 17,587,728 | 2,290,612,747 | 130.24 | 82.90 | ||
| PHS 2 | 15,576,512 | 2,124,079,990 | 136.36 | 86.64 | |
| 15,576,512 | 2,029,653,355 | 130.30 | 82.79 |
Table 4.
Statistics of reads mapping to the reference genome.
*Aligned 0 times : Number of reads that were not mapped to the reference gene sequence after pre-processing
*Aligned 1 time : Number of reads in which the number 1 mapping operates in the reference gene sequence after pre-processing
Table 5.
Up/down differentially integrative transcriptomic data from pre-harvest sprouting to control.
종자의 발아는 휴면성을 유도하는 호르몬인 ABA함량에 의해 결정되므로 수발아 조건에서 ABA의 함량 조절에 관련된 유전자들의 발현을 살펴볼 필요가 있다. 수발아 종자에서 선발된 DEGs를 KEGG의 mapper 프로그램을 사용하여 발현정도를 확인했다. ABA는 carotenoid생합성 과정 중 β-카로테인으로부터 합성된 네오산틴 (Neoxanthin)과 비올아크산틴 (Violaxanthin)이 제오신(Xanthoxin)으로 산화과정을 거쳐 ABA가 합성이 된다. ABA는 CYP707A (ABA 8’- Hydroxylase)에 의해 산화되어 비활성형인 Phaseic acid (PA)로 바뀐다. KEGG mapper 에서 제공되는 reference pathway보다 Oryza sativa japonica 품종의 carotenoid biosynthesis pathway에서 몇몇 유전자의 발현이 DEGs와 상관없이 낮게 발현되어 옅은 초록색으로 기본적으로 표시되었다(Fig. 2). 수발아 조건이 되면 ABA 생합성 및 분해 관여하는 유전자중 네오산틴과 비올아크산틴을 제오신으로 산화시키는 효소인 9-cis-epoxycarotenoid dioxygenase (NCED)의 유전자 발현이down-regulation되었다(Fig. 2). 벼에는 OsNCED1~ 5까지 5개의 NCED가 존재하며 발현 정도에 따라 종자 발아가 조절된다(Zhu et al., 2007). 또한 ABA를 비활성형인 PA로 산화시키는 CYP707A가 up-regulation되어 ABA 함량이 줄어들어 수발아가 일어나는 것으로 보인다(Fig. 2). 벼에는OsCYP707A1~A3까지 3개의 CYP707A효소가 존재하며NCED와 반대되는 역할로 ABA를 분해하여 종자 발아를 조절하는 것으로 알려져 있다(Zhu et al., 2007). OsNCEDs와 OsCYP707As 외에는 ABA 합성과 분해 과정에 직접적으로 관여하는 유전자의 유의미한 차이(1-fold change)는 RNA- sequencing 결과 나타나지 않았다. 이 결과를 바탕으로 수발아는 ABA 생합성 효소인 OsNCEDs의 발현 감소와 분해효소인 OsCYP707As의 발현 증가로 인함 ABA 감소로 발생하며 품종간 발현 차이로 인해 ABA의 감소 속도가 달라져 발아율에 차이가 생길 수 있다.
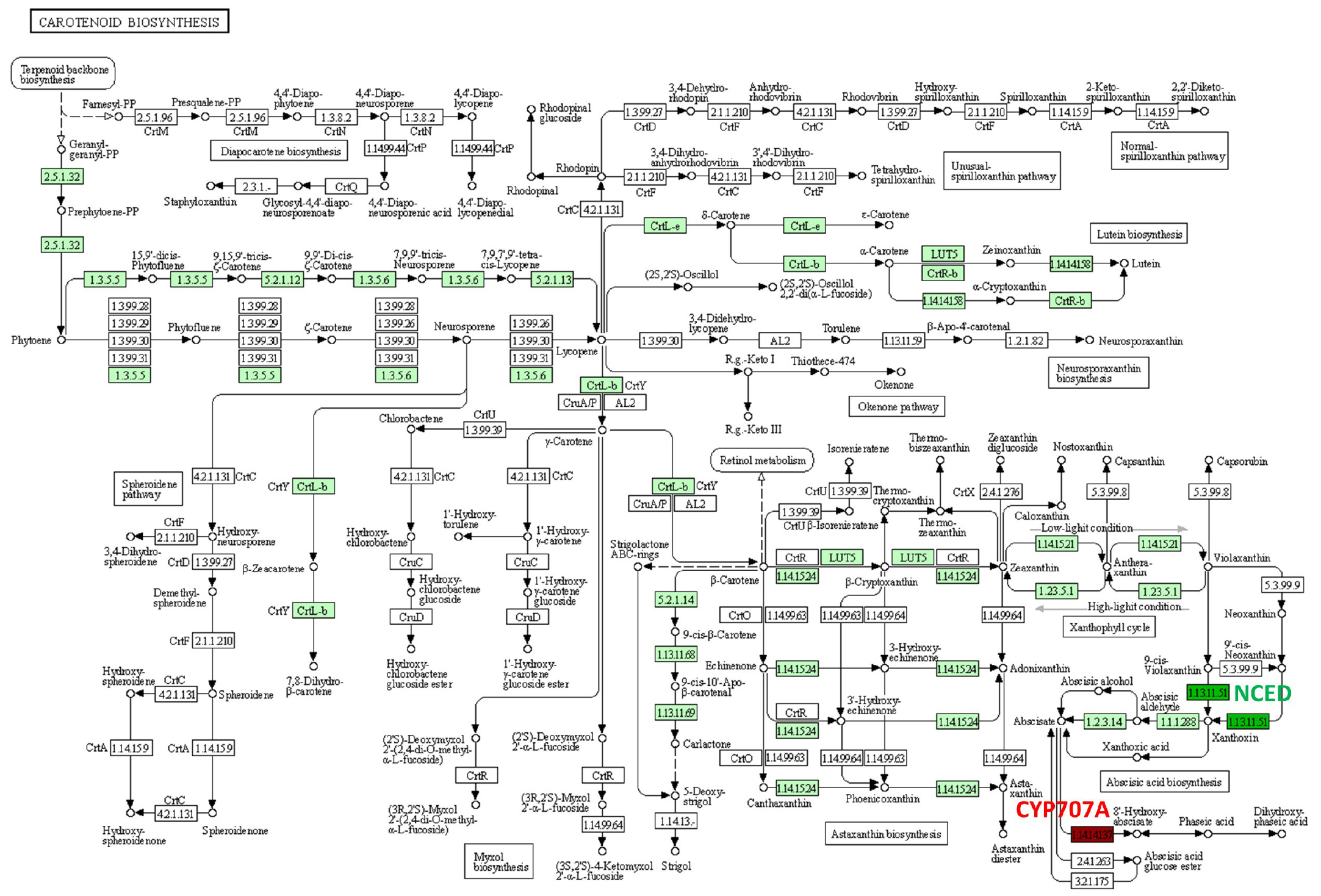
Fig. 2
Changes in the reaction related to carotenoid biosynthesis pathway in pre-harvest sprouting (PHS) seeds. The metabolism of each previously selected differentially expressed gene was analyzed using the KEGG mapper. Red and green indicate genes induced or suppressed by PHS, respectively. 1.13.11.51 : NCED; 9-cis-epoxycarotenoid dioxygenase, 1.14.14137 : CYP707A; (+)-abscisic acid 8'-hydroxylase.
RNA-sequencing 결과를 바탕으로 수발아 처리시 조평과 조운의OsNCEDs와 OsCYP707A의 발현정도를 quantitation real-time PCR을 통하여 확인하였다(Fig. 3). ABA 생합성 유전자인 OsNCED1과 OsNCED2, OsNCED3의 경우 유의미한 결과를 얻었으나 OsNCED4와 OsNCED5의 경우 발현량이 매우 낮아 결과에서 제외 하였다(no data). 대조군인 수발아를 처리 하지 않은 출수 후 적산온도 900°C일 때 종자에서 조평보다 조운의 OsNCED1~3가 높은 발현량을 보였다. 수발아 처리시 OsNCED1과 OsNCED2는 수발아 처리시 대조군 보다 두 품종 모두 발현량이 감소하였으며 온도가 높아질수록 발현량이 감소 하지만 조운이 조평보다 높은 발현량은 유지하였다. OsNCED3의 경우 조평은 전체적으로 발현량이 비슷하였으나 조운은 25/15°C에서는 조평보다 발현량이 높고 27/17°C와 27/29°C 처리시에는 조평보다 낮았다. OsCYP707A1은 수발아 처리시 발현량이 대조군 보다 높게 나타났으며 조평과 조운의 발현량 차이는 거의 없었다. OsCYP707A2는 온도가 높아짐에 따라 발현량이 낮아져 유의미한 결과를 나타내지 않았다. OsCYP707A3는 수발아 처리시 오히려 낮은 발현량을 보이는 구간이 있고 발형량차이가 크지 않아 유의미한 결과를 나타내지 않았다. 조평과 조운의 수발아율 차이가 ABA의 함량에 의해 결정된다면 OsNCEDs의 전체적인 발현차이로 결정될 것으로 보인다.
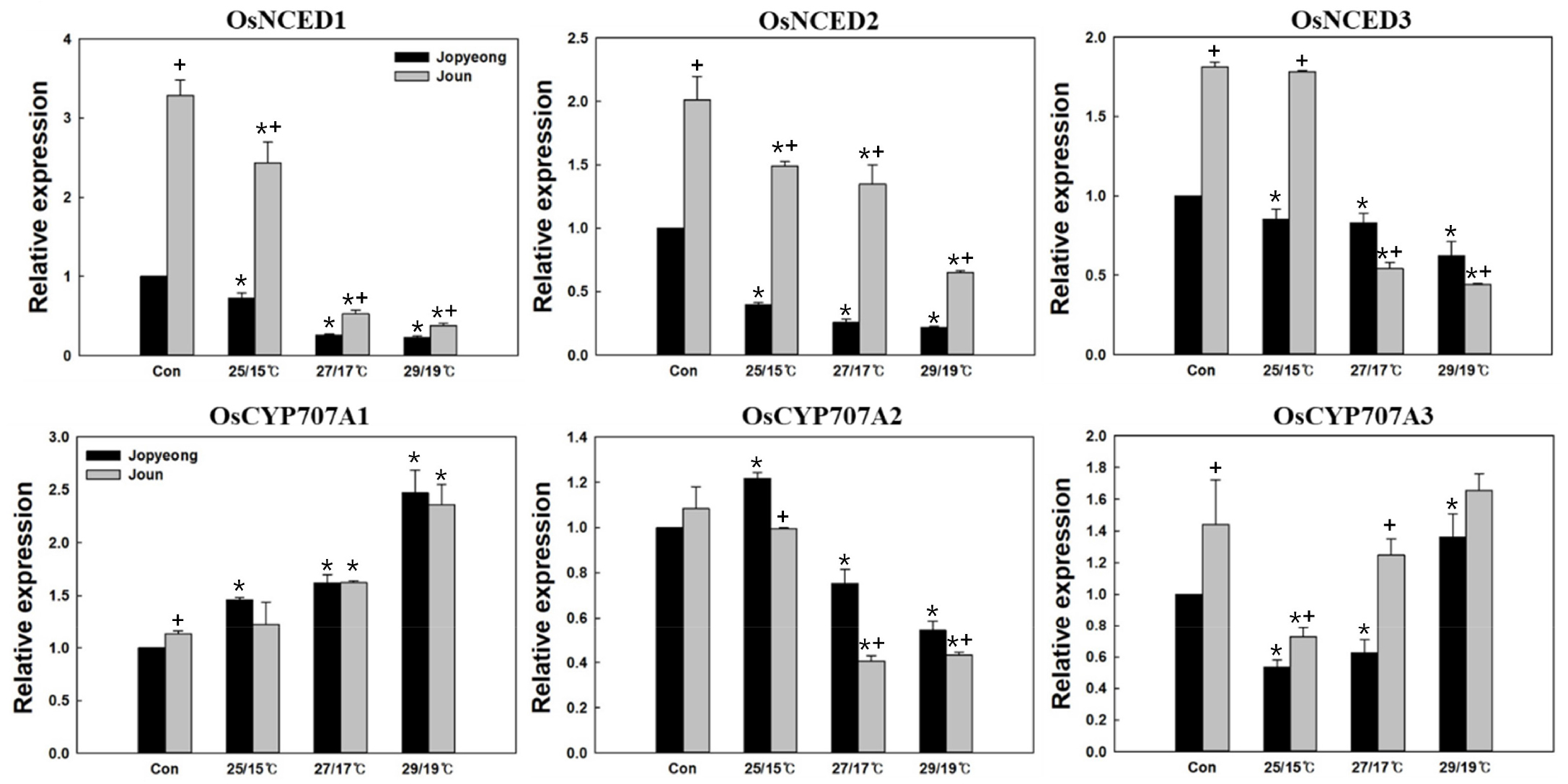
Fig. 3
Difference in relative expression of genes associated with abscisic acid (ABA) biosynthesis genes (OsNCEDs) and ABA catabolic genes (OsCYP707As). A 900°C Winkler scale after heading was used and treated for 3 days by test temperature. Data are mean ± standard error (* + p< 0.05, n = 3). *Control and different treatments within test temperature; + between Jopyeong and Joun.
벼 종자의 ABA 함량은 벼의 등숙 초기에서부터 증가하여 중기까지 증가하다가 점차 감소한다고 한다(Liu et al., 2014). 수발아가 시작될 때 ABA함량이 중요하다고 생각되어 수발아 처리 후 3일 된 종자의 ABA함량을 측정하였다. 조평과 조운 모두 등숙시 700°C 이후 등숙이 진행 될수록 종실내 ABA 함량이 감소하며 수발아 처리온도가 높을수록 ABA함량이 감소한다. 그러나 수발아 처리전부터 종실내 ABA함량이 조평보다 조운이 상대적으로 높으며, 수발아 처리시에서도 남아있는 ABA함량이 조운이 상대적으로 높다(Table 6). 이는 조평과 조운의 수발아성에 ABA함량이 상관관계가 있다고 할 수 있다. 등숙기간 상대적으로 높은 ABA 함량을 가지고 있는 조운이 수발아 처리시에도 아직 분해되지 않은 ABA함량이 높아 휴면성을 유지하여 수발아가 상대적으로 적게 나타난다. ABA함량과 수발아성의 관계는 조운과 조평처럼 수발아 처리전 ABA함량에 따라 결정될 수 있지만 수발아 처리전 ABA함량이 높아도 상대적으로 수발아성이 높은 품종이 존재한다(Kang et al., 2018). 조평과 조운의 경우 수발아성 차이가 OsNCEDs의 발현량 차이로 인한 ABA 함량 차이로 보이지만 품종마다 ABA의해 결정되는 종자의 휴면성에 대한 민감도가 다를 수 있고, 또 다른ABA생합성 유전자와 ABA 분해유전자의 발현 차이로 인한 ABA 함량 변화 속도 또한 수발아성을 결정할 것으로 생각된다.
Table 6.
Abscisic acid content in rice cultivars according to the Winkler scale after heading and test temperature.
적 요
본 연구는 조평과 조운의 벼 출수 후 적산온도와 검정온도에 따른 수발아 발생 정도를 조사하고, RNA-sequencing 방법을 사용하여 수발아 발생 원인을 찾았다. 품종간 수발아성에 관여하는 생리적, 유전학적 요인을 구명하고자 수행하였으며 분석한 결과는 다음과 같다.
1. 출수 후 적산온도가 높아질수록, 검정온도가 높아질수록 수발아 처리시 수발아율이 높았고, 조운벼가 내수발아성이 강하고, 조평벼가 수발아성이 높은것으로 나타났다.
2. 수발아성이 높은 조평벼를 대상으로 한 RNA-sequencing 결과 ABA 생합성에 관여하는 OsNCEDs의 발현이 감소하고, ABA 분해에 관여하는 OsCYP707As의 발현이 증가하였다.
3. 조평과 조운의 OsNCEDs와 OsCYPY707As의 Quantitation Real-Time PCR 결과 조평보다 조운에서 OsNCEDs의 발현이 높게 나타나 수발성과 상관관계를 보였으나, OsCY P707As는 수발아성과 상관관계를 보이지 않았다.
4. 조운벼는 등숙기간중 종실내 ABA함량이 조평보다 높으며 수발아 처리시 남아있는 ABA함량이 높아 내수발아성이 상대적으로 강하게 나타났다.