서 론
화본과(Poaceae family)의 진화
BOP 클레이드
PACMAD 클레이드
포아풀아과(Pooideae subfamily)의 진화
포아풀아과의 비교유전체 연구
포아풀아과의 진화과정: 기후변화, 서식지의 이동
포아풀아과(Pooideae subfamily)의 육종: 맥류 및 한지형 목초 중심의 고찰
미래연구방향
결 론
서 론
화본과(Poaceae)는 피자식물 중 가장 큰 식물과로서 지구 표면의 약 20%를 차지하고 있으며 진화적으로 또는 생태적으로 가치가 매우 높은 식물들로 이루어져 있다. 또한 화본과는 경제적으로도 매우 중요한 작물들이 많이 포함되어 있으며 대나무, 목초, 식량작물을 포함하여 약 700여속 10,000여종으로 구성되어 있다. 이러한 화본과는 약 1억 5천만년전에 분화가 일어났으며 C3와 C4 광합성 시스템을 대표하는 BOP와 PACMAD 클레이드로 구성이 되어있다. 화본과(Poaceae) 식물은 약 1억 5천만년전 쌍떡잎식물에서 분리되어 독립적으로 진화해 왔으며 (Chaw et al., 2004) 단일 식물과로서 가장 큰 위치를 차지하기 때문에 진화적으로 매우 가치 있는 식물종이 다양하게 포함되어 있다. 특히 주요 진화 요인 중의 하나인 전체 유전체 중복(whole genome duplication)이 다양한 모델 식물의 유전체 분석을 통해서 밝혀지면서 화본과 식물 특이적인 유전체 구조의 변화에 관련된 연구와 함께 분류학적인 연구도 활발하게 진행이 되고 있다. 그 중에서도 가장 많은 종을 보유하고 있는 포아풀아과는 온대 및 한대지역에서 광범위하게 적응하고 있는 식물들로서 시신세(Eocene)에서 점신세(Oligocene) 사이(시신세-점신세 전환기, E-O transition, 약 3,400만년전)에 북반구를 중심으로 지구의 대기온도가 하강하면서 급속도로 분화된 것으로 알려져 있다(Favre et al., 2016; Meseguer et al., 2018). 그러나 지구상 종자 식물과의 80% 정도가 열대지역에 서식하며 약 20%정도의 식물과가 한대지역에 서식하는 식물종을 포함하고 있다는 사실 (Stevens, 2001)에 기초할 때 열대식물에서 한대식물로의 전이 과정은 간단하지 않을 것으로 생각이 되고 있으나 한대지역에 서식하는 식물종의 다양성과 진화 연구의 어려움으로 인하여 많은 부분이 밝혀지지 않고 있다.
포아풀아과(Pooideae subfamlily)는 거의 4,000여종의 식물을 포함하는 화본과 중 가장 큰 아과이며 맥류를 포함한 다양한 경제적 작물들이 포함되어 있다(Soreng et al., 2017). 포아풀아과는 한지형 화본과 식물을 대표하며 추위에 견디는 형질을 중심으로 진화를 거듭해 온 결과 지구상에 가장 추운 지역에서 자랄 수 있는 적응력을 갖게 되었으며 이러한 능력은 포아풀아과의 모델 식물로 자리잡은 Brachypodium distachyon (Bracypodieae 족)과 핵심 포아풀아과(core Pooideae)의 연구로 많은 부분이 밝혀지고 있다. 핵심 포아풀아과는 약 14개의 족(tribe)에 포함되어 있는 약 3,232종으로 구성되어 있으며 (Soreng et al., 2017), 포아풀아과에서 경제적으로 중요한 작물인 빵밀(Triticum aestivum), 보리 (Hordeum vulgare), 한지형 잔디 또는 사료작물[블루그래스(Poa spp.), 벤트그래스(Agrostis spp), 페스큐(Festuca spp.), 라이그래스(Lolium spp.)]이 포함되어 있다. 핵심 포아풀아과는 다른 포아풀아과의 족(tribe)들보다 상대적으로 가장 늦게 분화가 되었으며 몇가지 종들을 제외하고는 7개의 기본염색체 수를 가지고 있고 배수성인 식물이 우점한다(Pimentel et al., 2017). 포아풀아과는 열대 및 아열대지역에 서식하는 분지군벼아과(Oryzoideae)및 대나무아과(Bambusoideae)와 공통의 조상을 가지며 BOP 클레이드를 형성하고 이는 C3 광합성계를 가지는 화본과 아과들을 대표하고 있다. 따라서 추운 기후에 적응하며 진화해온 과정은 포아풀아과를 연구함으로서 C3 광합성계의 진화과정 및 BOP 클레이드의 형성 과정 등에 관련된 이해도를 넓힐 수 있다(Schubert et al., 2018).
포아풀아과의 기원은 4,500만년에서 6,400만년 전으로 알려져 있으나(Prasad et al., 2011; Wang et al., 2015) 약 2,000만년의 시간차는 포아풀아과의 정확한 기원을 정의하기에는 무리가 있다. 이러한 시간차는 포아풀아과의 기원에 관련한 화석자료 등의 부재 또는 화본과 식물이 다른 피자식물보다는 느린 염기서열 변화 속도 등으로 인하여 그 진화계통도(phylogenetic tree)가 확실하게 밝혀져 있지 않은데 기인한다(Schubert et al., 2019; Pimentel et al., 2017). 따라서 많은 연구들이 신생대의 기후변화와 포아풀아과의 분화와 관련지어 진행되고 있다.
본 총설에서는 화본과에서 한대식물을 대표하고 있으며 경제적으로 중요한 작물을 다수 포함하고 있는 포아풀아과의 한대지역 적응성과 관련된 기원과 진화, 그리고 맥류를 중심으로 한 육종을 위한 자원 및 과정 등을 고찰하기 위하여 작성되었다.
화본과(Poaceae family)의 진화
화본과 식물(Poaceae)은 경제적, 생태학적으로 중요한 역할을 하는 현화식물에서 가장 큰 식물군 중 하나이며, 약 700속과 10,000여 종을 포함한다(Zhao et al., 2013). 화본과 식물은 1억 4천 - 1억 5천만년 전(쥐라기 말-백악기 초기)에 쌍떡잎식물에서 분리되었고, 약 6,500만년 전 칙술루브 소행성 충돌 직후(Cretaceous-Paleogene 또는 K-Pg boundary) 전체 유전체 중복(whole genome duplication) 과정을 거친 이래로 독립적으로 진화된 것으로 받아들여지고 있다(Chaw et al., 2004). 화본과 식물의 아과는 크게는 아과명의 머리글자를 따서 BOP 클레이드와 PACMAD 클레이드로 나누어지며 두 클레이드가 분화되기 이전에 독립적으로 진화된 것으로 생각되는 아노모클로아아과(Anomochlooideae), 파루스아과(Pharoideae), 푸엘리아아과(Puelioideae)로 구성되어 있다(Fig. 1). 가장 먼저 분화한 혈통인 아노모클로아아과와 파루스아과는 각각 7천6백만년과 7천4백만년 전에, 푸엘리아아과는 5천8백만년 전에 발생한 것으로 추정된다. BOP와 PACMAD 클레이드의 기원 시점(crown node)은 초기 에오세인 5천7백만년 전인 것으로 추정되었다(Bouchenak- Khelladi et al., 2010). 그러나 최근 연구에 따르면 PACMAD 클레이드의 crown node에서 다양성을 야기한 분화 시점은 3,240만년 전으로 추정되고, 이는 신생대의 시신세-점신세 전환기(E-O transition)와 일치하는 것으로 나타났다(Cotton et al., 2015). BOP와 PACMAD 클레이드는 각각 C3 및 C4 광합성 시스템을 나타내어 이들의 진화 역사 동안 기후 변화와 관련된 광합성 시스템을 연구할 수 있다. 이 섹션에서는 화본과 진화의 큰 줄기인 BOP 클레이드와 PACMAD 클레이드의 진화에 대해 간략하게 설명한다.
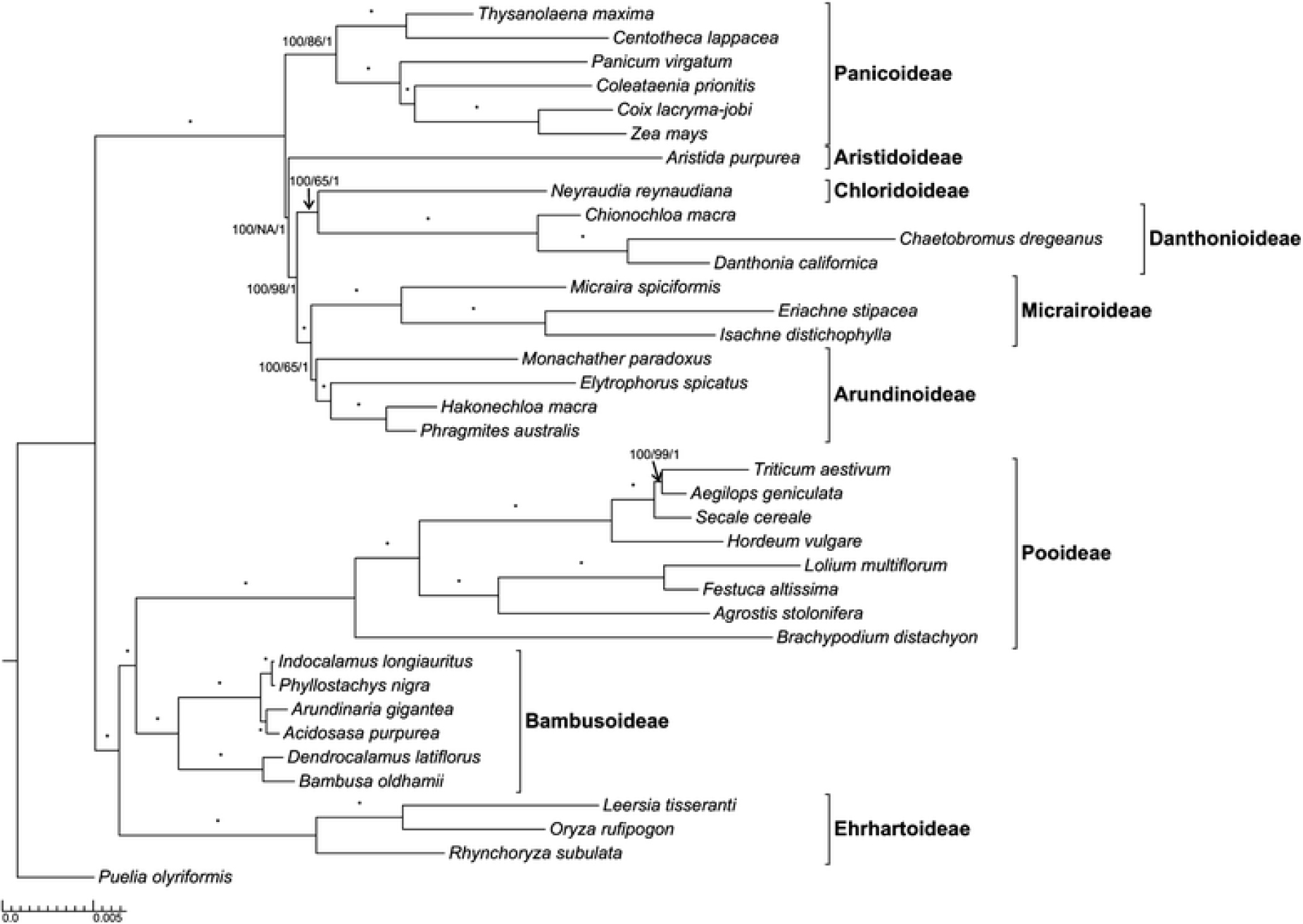
Fig. 1.
The phylogenetic tree of the BOP and PACMAD clades of Poaceae (Cotton et al., 2015). The indicated numbers represent the maximum likelihood (ML) boostrap (bs)/ maximum parsimony (MP) bs value/ posterior probability (pp) produced from a complete plastome analysis. Each node that was fully supported with a ML bs value = 100, MP bs value = 100, and pp = 100 is marked with *.
BOP 클레이드
BOP 클레이드는 대나무아과(Bambusoideae), 분지군벼아과(Oryzoideae) 및 포아풀아과(Pooideae)의 세 가지 아과로 구성되어 있으며, 이들은 모두 C3 식물로 이루어져 있다(Christin & Osborne, 2013). BOP 클레이드 중 첫 번째 분지인 BOP 분지군벼아과는 5천3백만년 전(시신세 중기)에 유래되었으며, 19속과 115종을 포함한다. 대나무아과는 125속 1,670종이 분포하고 2천9백만년 전에 분화된 것으로 추정되며, 온대 대나무(Arundinarieae), 열대 대나무(Bambuseae) 및 초본성 대나무(Olyreae)의 세 가지 족(tribe)으로 나뉜다. 이들은 각각 1060만년 전, 1930-1960만년 전, 그리고 2230만년 전에 분화된 것으로 알려져 있다(Ruiz-Sanchez, 2011). 포아풀아과는 대나무아과와 자매 그룹이며, 화본과에서 가장 큰 아과로 15족 202속 3,968종을 포함한다(II, 2012). Bayesian clock을 이용한 분자 데이팅 분석 결과 4천4백만년 전에 유래된 것으로 알려졌으나 문헌에 따라 차이가 큰 편이기 때문에 화석 연구 및 모델 식물의 확대를 통한 추가 연구가 필요한 실정이다(Bouchenak- Khelladi et al., 2010; Soreng et al., 2017).
PACMAD 클레이드
PACMAD 클레이드는 PACMAD 분지군아리스티다아과(Aristidoideae), 기장아과(Panicoideae), 물대아과(Arundinoideae), 미크라이라아과(Micrairoideae), 단토니아아과(Danthonioideae) 및 나도바랭이아과(Chloridoideae)로 구성되어 있다. 모든 종이 C3로 이루어진 BOP 클레이드와 달리 PACMAD 클레이드는 대부분 C4 식물로 구성되며, 두 클레이드의 분화 후에 진화한 일부 C3 식물도 포함하고 있다(Christin & Osborne, 2013). PACMAD의 분지군 아리스티다아과는 2천8백만년 전에 유래되었으며, Aristida (250- 290종), Stipagrostis (50종, 아프리카-아시아) 및 Sartidia (5종, 아프리카 및 마다가스카르)의 3가지 속이 있다(Cerros- Tlatilpa et al., 2011). 이 중 Aristida와 Stipagrostis는 거의 C4 종으로만 구성되는 반면, Sartidia는 작은 C3 그룹으로 이루어져 다른 족들과 확연히 구분이 된다(Christin & Besnard, 2009). 기장아과는 벼과에서 두 번째로 큰 아과이며 수수, 사탕수수, 옥수수 등 경제적으로 중요한 작물들을 포함한 212속 3,316종으로 구성되어 있다(Burke et al., 2016). 기장아과는 PACMAD 조상과 유사하게 나무가 없고, 평야, 툰드라 지역 같이 바람, 빛, 비에 많이 노출되는 열린 서식지와 그와 반대인 닫힌 서식지가 혼합되어 있어 약 2천4백만년 전(초기 중신세)에 발생한 것으로 추정했으나, 최근의 연구에서는 초기 분지가 약 3천8백만년 전인 것으로 나타났다(Bouchenak-Khelladi et al., 2010; Zeng et al., 2012). 물대아과는 2족 14속 40종을 포함하며, PACMAD 클레이드에 속하지만 모두 C3 식물로 구성되어 있다(Soreng et al., 2017). 물대아과는 형태학, 생태학적으로 좁고 깊은 속에 속하기 때문에 화본과에서 해결되지 않은 분류학적 미스터리로 여겨지며 향후 광합성 체계와 관련된 추가 연구가 필요한 아과이다(Christin et al., 2014). 물대아과와 자매 그룹인 미크라이라아과는 4족 186종이 분포한다(II, 2012; Soreng et al., 2015) 미크라이라아과는 C4 광합성과 관련하여 일반적인 경향과 반대의 모습을 보인다(Spriggs et al., 2014). 같은 PACMAD 클레이드에 속한 식물들은 C4 식물이 평균적으로 C3 식물에 비해 높은 수준의 다양화를 보이는데, 미크라이라아과에서는 C4 식물로 구성된 족인 Eriachneae보다 C3 식물로 구성된 Isachneae이 더 넓은 서식지 유형을 보이며, 더 많은 종을 포함한다. Isachneae의 일부 C3 종은 진화과정에서 그늘진 서식지로의 전환에 따라 다변화를 겪은 것으로 생각되고 있다(Teisher, 2016). 단토니아아과는 1족 19속 292종이 분포하며, PACMAD 클레이드에 속하지만 C3 광합성 경로만을 사용한다(II, 2012; Soreng et al., 2017). 단토니아아과는 주로 남반구 화본과의 아과이며, 분화 시점은 약 3천8백-2천1백만년 전으로 추정한다(Bouchenak- Khelladi et al., 2010). 나도바랭이아과는 단토니아아과와 자매 그룹으로, 대부분의 종(일부 종 제외)이 C4 광합성 경로를 사용한다. PACMAD 클레이드에서 기장아과 다음으로 큰 아과이며, 131속 1,601종으로 구성된다(Soreng et al., 2015). 가장 오래된 C4 식물의 기원이 나도바랭이아과(3천2백-2천5백만년 전)에서 발견되어, 이를 통해 최초의 C4 광합성의 기원을 알 수 있다(Christin et al., 2008).
포아풀아과(Pooideae subfamily)의 진화
포아풀아과(Pooideae)의 기원은 중생대 후기 백악기(Cretaceous)에서 신생대의 초기 고신세(Palaeocene) 기간 중이지만 이 당시의 기후는 온화하고 저위도와 고위도 간 온도 차이가 크지 않았으나(Mudelsee et al., 2014; Zachos., 2001), 시신세에서 점신세(Eocene-Oligocene) 동안 온도가 낮아지는 기후 변화가 진행되었다(Schubert et al., 2019c). 따라서 포아풀아과의 본격적인 분화는 후기 시신세(Eocene)에서 시작 되었을 것으로 예상이 되며, 점신세에서 중신세(Oligocene-Miocene)동안 매우 활발하게 일어난 것으로 추정된다(Pimentel et al., 2017). 그러나 대기의 온도가 떨어지는 과정이 어떻게 포아풀아과 진화에 영향을 주었는지는 포아풀아과의 전체적인 phylogeny와 정확한 분지 시점의 연구가 선행되지 않으면 밝혀내기는 어려울 것으로 생각이 된다. 하지만 2800만년전 CO2가 풍부한 환경에서 대부분의 C3 광합성을 하던 식물들이 기후 변화와 대기중 CO2가 감소함에 따라 진화하여 C4 광합성을 하는 식물로 진화했고, C3 식물들이 넓어진 온대기후 식생으로 옮겨 갔을 것이라는 합리적인 추론은 가능하다. C4 식물의 광합성은 온도가 높고 건조한(물 부족) 지역인 적도와 사바나 지역에서 잘 자라며 C3 식물의 광합성은 비교적 온도가 낮은 지역에서 이루어진다(Edwards et al., 2010). 하지만 고대 온도 변화가 C3 식물에서 C4 식물로 진화하는 과정과 정확하게 일치하지는 않으며, C4 종 일부 진화과정만이 매년 감소하는 평균 강수량과 일치하는 경향을 보인다. 이는 C4 식물의 서식지 이동이 기후변화에 따른 진화 과정이라는 측면 보다는 C3 식물의 내한성이 목초지 변화 및 이동에 영향을 주었다는 사실이 진화적 중요 요인이라는 것이다(Edwards & Smith, 2010). 그러나 화석 기록, 분자 연대 측정 결과, 기후변화 자료 간의 불일치는, 관련된 데이터들의 부재와 분화가 일정한 시간 동안에 발생하지 않았으며, 또한 여전히 많은 연구자들은 포아풀아과가 C4식물로 출현하여 C3 식물로 진화하였는지의 진위 여부와 포아풀아과가 온대기후로 어떻게 전이 하였는지, 그리고 중생대의 기후변화 등이 포아풀아과의 진화와 매우 관련이 클 것으로 생각하고 있다(Schubert et al., 2019). 열대성 C4 식물이 온대성 C3 식물로 분화되어 서식지를 온화한 기후대로 옮겨가는 과정 중 동계의 낮은 온도 환경에서 살아남기 위해 당 함량 증가, 막의 지질 구성 변화, 내동성 단백질 합성 등 생리적 변화로 인한 내동성(freezing tolerance)이 증대되는 새로운 형질을 얻게 되었는데, 이 과정을 저온 순화(cold acclimation)라고 한다(Schubert et al., 2019a). 포아풀아과의 내동성 특징은 단기간, 장기간 저온에 의해 발현이 유도되는 유전자가 뚜렷하다. 이러한 저온 발현 유도 유전자(cold-induced genes)는 저온에 대한 노출이 단기간일 때 상향 조절되고, 장기간 일 때 하향 조절되면서 추운 환경에 적응할 수 있다. 따라서 포아풀아과의 주요 서식지가 열대지역에서 온대 지역으로 전이된 중요한 요인은 내동성 때문이라고 할 수 있을 것이다(Zhong et al., 2018).
포아풀아과의 비교유전체 연구
포아풀아과의 진화과정: 기후변화, 서식지의 이동
핵심 포아풀아과(Core Pooideae)는 약 14개의 족(tribe)에 포함되어 있는 약 3,232종으로 구성되어 있으며(Soreng et al., 2017) 경제적으로 중요한 작물인 밀(Triticum aestivum), 보리(Hordeum vulgare), 귀리(Avena sativa), 라이그래스(Lolium perenne), 한지형 잔디류(이용가치 상 사료작물로 분류)(Agrostis spp., Poa spp., Lolium spp., Festuca spp.)등이 포함되어있다(Schneider et al., 2011). 식물의 유리한 환경 조건과 번식량을 일치시키는 능력은 식물의 일생에 있어서 중요한 영향을 미친다(McKeown et al., 2016). 이때, 온대 생태계에서 핵심 포아풀아과를 비롯한 몇몇 식물종들은 겨울에 얼지 않을 정도의 추위에서 장시간 노출된 후(춘화, vernalization) 이듬해 봄에 온화한 기후에서 생식성장으로 전환하는 능력을 진화시켜 봄의 개화를 준비한다(Chouard, 1960; Heide, 1994; McKeown et al., 2016). 뿐만 아니라, 핵심 포아풀아과의 조상은 짧은 기간 동안 낮은 온도 노출로 인하여 내동성으로 추론되는 저온 순화(cold acclimation) 과정을 통해 추위에 의한 기계 및 생리학적 세포 손상에서 살아 남았다(Thomashow, 1999). 하지만 춘화에 의한 개화와 저온 순화의 유전은 포아풀아과와 관련이 먼 모델 종에 의해 설명되었고, 각각의 종은 복합체 형태의 많은 유전자와 상호간의 연결된 유전자 의해 조절된다(Deng et al., 2015; Dhillon et al., 2017; Kippes et al., 2015; Thomashow, 1999; Yan et al., 2006; Yan et al., 2004; Yan et al., 2003; Zhao et al., 2015).
Pooideae 중 곡식류에서는 춘화 반응은 3개의 핵심 단백질인 VERNALIZATION1 (VRN1), VRN2 and VRN3 의해 조절된다(Kippes et al., 2015; Yan et al., 2006; Yan et al., 2004; Yan et al., 2003). 저온 순화는 C-REPEAT BINDING FACTOR/DEHYDRATION-RESPONSIVE ELEMENT BINDING PROTEIN (CBF/DREB)과 Cold-regulated (COR) 유전자 수와 연관된 변화에 의해 조절이 된다(Fowler & Thomashow, 2002; Thomashow, 1999; Zhao et al., 2015). CBF 유전자는 저온 스트레스를 빠르게 감소시켜 주는 유전자로 잘 알려져 있다(Medina et al., 1999). CBF 유전자는 관련이 먼 잔디 계통에서 급속한 저온 유도에서 보전된다는 관찰이 이루어졌다. 이 유전자는 단기적인 추위에 노출된 저온 순화와 저온 유발 스트레스 반응의 진화가 장기적인 저온 반응의 진화보다 먼저 발생하였다. 장기적인 저온 반응은 서리 저항성과 개화 능력으로 전이되었다(Zhong et al., 2018). 단기적 추위에서 장기적 추위로의 내한성 전이는 계통 특이적 복제를 하며, 그에 따라 새롭거나 분할된 유전자가 수반될 것이다(SANDVE & Fjellheim, 2010; Tondelli et al., 2011). 포아풀아과는 기원에서부터 현재까지 시간이 흐르면서 분화율이 변화하였는데, 신생대의 Eocene부터 Miocene (약 5580만년 전부터 약 600만년 전)동안 지구의 온도가 약 17°C에서 약 5°C까지 낮아지는 동안 Fig. 2의 분화율 신뢰구간 변화를 통해 포아풀아과의 분화율이 점차 증가한 것을 알 수 있다(Schubert et al., 2019, Fig. 2). 그리고 추위와 분화 간의 연관성에서 가능한 원동력은, 신생대 중반에서 후반까지 온대기후가 Holarctic (전북구)로 크게 확대되었고(Damuth et al., 1992; Eldrett et al., 2009; Liu et al., 2009; Pound & Salzmann, 2017; Zachos et al., 2001), 서리로 인해 대부분의 열대 목질 종들이 제거되어 내한성인 풀들의 서식지가 늘어나고 경쟁이 줄어들었다(Schubert et al., 2019c). 또한, 추위를 견디기 위한 사전 적응(Pre-adaptation)은 포아풀아과가 온대기후로 확장되고 계속해서 더 추운 환경에 적응함으로써 분화율이 증가한 것을 설명할 수 있다(Schubert et al., 2019c). 하지만 결론적으로 포아풀아과에서 추위-반응성 유전자가 어떻게/언제 기원 했는지 정확히 알 수 없으며, 포아풀아과의 분화 동안 이루어진 진화에 대한 진화론적 견해가 많이 부족하고 정확히 알려지지 않아 내한성이 생긴 원인을 단정 지을 수 없다(Zhong et al., 2018).
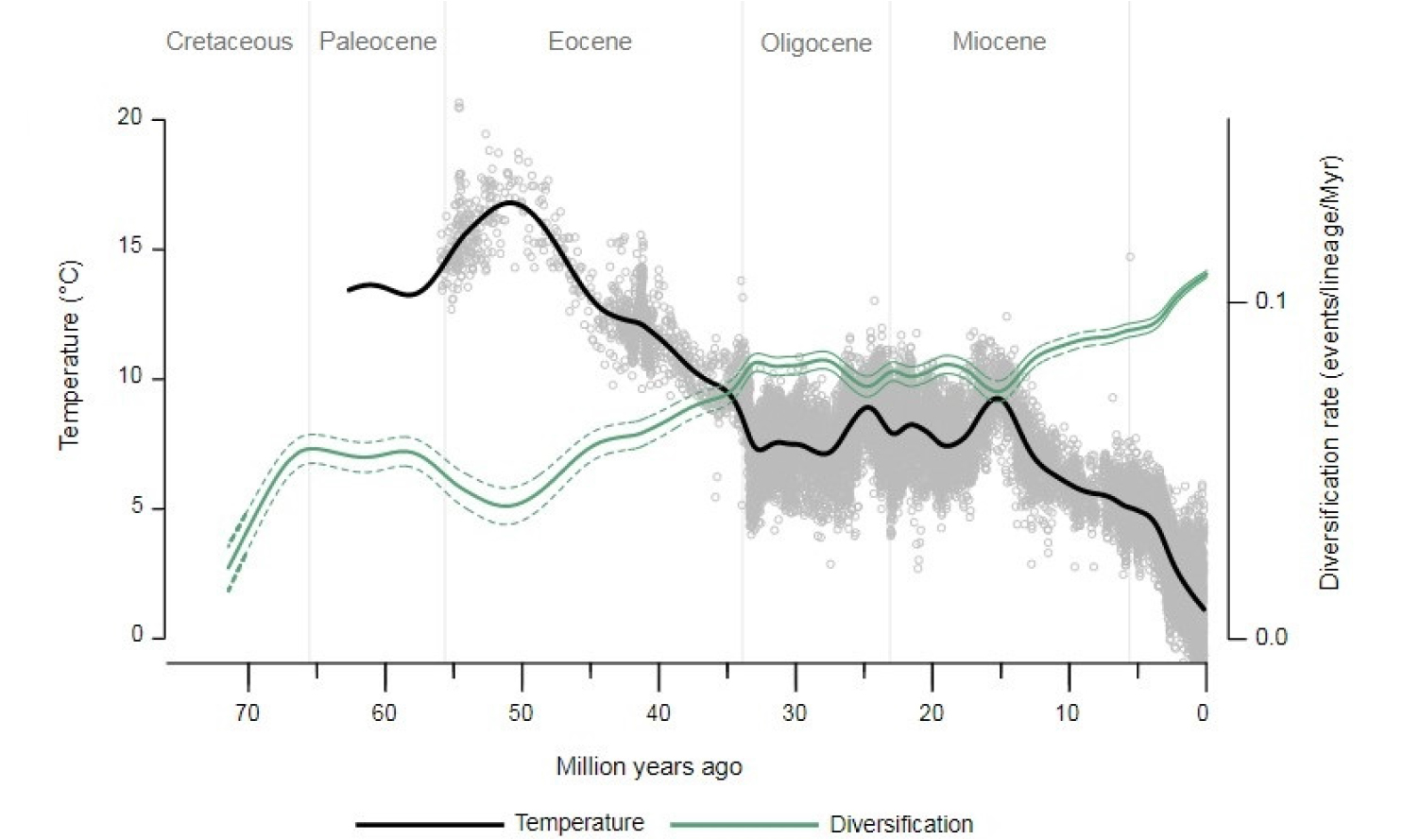
Fig. 2.
Palaeotemperature-dependent diversification model: diversification rates in Pooideae (in green) exhibited an increasing trend with the decreasing global temperatures (in black). The temperature is represented by the global mean temperature curve from the Palaeocene period to the present, modified from Zachos, Dickens, and Zeebe (2008). The temperature curve should be treated with caution, because it is computed assuming an ice-free ocean (for details, refer to Zachos et al., 2008). Dashed green lines represent confidence intervals of the estimated values from 100 phylogenetic trees randomly selected from the posterior distribution (Schubert et al., 2019b).
포아풀아과(Pooideae subfamily)의 육종: 맥류 및 한지형 목초 중심의 고찰
약 12,000년 전, 소수의 인간 집단이 사냥 및 채집을 통한 생활에서 지속적인 생존을 위해 농사를 시작하였고 이후 농업의 발전은 Neolithic revolution으로, 인간 문화에 혁신적인 변화를 일으켰다(Childe, 1953). 농업을 개척한 최초의 인류는 Fertile Crescent (비옥한 초승달 지역; 현대 이스라엘, 요르단, 레바논, 시리아 서부에 걸쳐 터키 남동부까지, 그리고 티그리스 강과 유프라테스 강을 따라 이라크와 이란의 서부)에 거주하며 문명을 발전시켰다(Gopher et al., 2001; Moore et al., 2000). 초승달 지역에서 현재 곡식들의 조상들인 야생 밀(Triticum urartu, T. boeoticum and T. dicoccoides), 야생 보리(Hordeum spontaneum), 야생 호밀(Secale vavilovii)이 등장하였고, 야생 종의 종자들은 고고학적 유적지에서 방사성 탄소와 지층의 연속성을 통해 밝혀졌다(Gopher et al., 2001; Moore et al., 2000).
또한, 작물의 수확량 향상, 생물학적, 비생물학적 스트레스 내성, 품종의 우수한 형질들은 모두 주된 육종의 목표이며, 이처럼 작물 개선에 필수적인 육종에 가치 있는 새로운 형질 탐색과 유전적 다양성을 보존하기 위해 생식질 수집은 중요한 자원이다. 작물 육종을 위한 이러한 유전자원의 활용도를 극대화시키기 위해서 생식질들의 모집단을 대변할 수 있고, 특성 평가가 충분히 이루어진 소집단이 필요하며, 이것을 ‘핵심집단(Core collection)’이라고 한다(Frankel et al., 1984; Brown et al., 1989; Wang et al., 2018).
밀
야생 종 T. boeoticum에서 순화된 2배체 Einkorn 밀(Triticum monococcum; AA genome)은 최초로 경작된 밀이다(Harlan & Zohary, 1966; Van Zeist et al., 1991). 하지만 청동기 시대부터 Einkorn 밀의 경작이 점차 줄어들었다(Nesbitt & Samuel, 1995). Einkorn 밀의 순화는 Einkorn 밀과 야생 조상 종인 T. boeoticum의 288개 유전자좌를 Amplified Fragment Length Polymorphism (AFLP) 마커를 통해 확인하였다(Heun et al., 1997). 현재 다배체 밀의 진화과정에서 중요한 단계인 4배체 emmer 밀(T. dicoccum)의 야생 종 4배체인 T. dicoccoides (AABB genome)으로부터 순화되었다(Simonetti et al., 1999). 야생 종 emmer 밀(T. dicoccoides; AABB)은 T. urartu로부터 AA genome을 받은 것으로 알려져 있다(Dvorak et al., 1998; Dvořák et al., 1993). 터키 남동부로부터 온 야생 emmer 밀(T. dicoccoides; AABB)과 순화된 4배체 밀(T. dicoccum; AABB)은 AFLP 분석을 통해 나온 204개의 유전자좌를 통해 서로 밀접한 관련이 있음을 확인하였다(Moore et al., 2000). T. dicoccum (AABB)에서 T. durum (AABB), T. turgidum (AABB)이 차례로 순화되었으며 4배체(AABB)인 T. turgidum (AABB)과 야생의 Aegilops tauschii (DD)가 알려지지 않은 시간과 공간에서 잡종화되어 6배체 밀인 T.aestivum (AABBDD)가 발생하게 되었다. 야생의 4배체 밀인 T. dicoccoides (AABB)은 Ae.tauschii (DD)와 분자유전학 적으로 겹치는 부분이 없어서, 순화된 T.turgidum (AABB)의 개입이 주요한 가설로 여겨지고 있다(Salamini et al., 2002).
이렇듯 다양한 잡종화에 의해 밀의 품종들은 유전적으로 다양성을 갖게 되고 다배체화 되었기 때문에 재배를 하기 위해서는 이 유전적 다양성을 형질 및 특성에 따라 이용할 수 있도록 핵심집단을 구축하여 품종 선택에 있어 유용하게 사용되어진다. Triticum aestivum L.의 핵심집단은 genomic- SSR과 EST-SSR 분자 마커들을 이용하여 지질학적 기원에 따라서 3942 계통에서 372계통으로 구축이 되었으며(Balfourier et al., 2007), 또한 생태학적 위치(유전적 다양성에 따른 차이)와 21개의 농업 및 식물학적 특징들을 SSR마커를 이용하여 23090계통에서 5029계통의 핵심집단을 만들었다(Hao et al., 2008). 최근 국내에서는 차세대바이오그린21 사업을 통하여 한국형 밀 핵심집단이 구축되어 다양한 연구가 진행중에 있다(Choi et al., 2018).
보리
보리는 아주 오래된 작물이며 10,000년 전에 근동 지역 최초의 순화된 작물 중 하나이다(Robinson, 2013; Zohary & Hopf, 2000). Fertile Crescent (Zohary & Hopf, 2000)의 여러 위치에서 발견된 보리의 고고학적 유적은 작물이 기원전 8000년경에 순화되었음을 나타낸다. 그리고 SSR 마커를 통해 2조종 보리와 6조종 보리가 각각 야생 2조종 보리인 Hordeum spontaneum과 야생 6조종 보리인 Hordeumagriocrithon을 통해 다른 야생원종에서 기원 되었다는 것이 밝혀졌으며, 현재까지 가장 유력하게 받아들여지고 있다(Jilal et al., 2008). 야생 보리의 대부분의 유전적 다양성은 중동에서 성장하는 개체군에 집중되어 있다는 여러 증거가 발견되었다(Nevo, 1992). 순화된 보리와 비교하여 야생형 보리는 더 좁은 잎, 약간 작은 종자, 긴 줄기, 그리고 완전히 성숙하기 전 수확했을 때 덜 약한 꽃대를 가지며(Anderson, 1992), 순화된 형태의 보리는 종자중이 증가하였고, 6조종 보리가 출현하였다(Salamini et al., 2002). 그리고 소수의 순화된 보리는 염색체 1번에 존재하는 열성 대립인자인 Naked caryopsis (N)유전자가 존재하기 때문에 나출종자를 갖고 있다(Søgaard & von Wettstein-Knowles, 1987). 또한 종자의 휴면성은 자연에서 야생 계통의 지속성을 증가시키는 긍정적인 요소인데(Li & Foley, 1997), 순화과정에서 종자의 휴면성 감소가 선택되었다(Salamini et al., 2002).
적절한 육종 프로그램을 시작하려면 이용가능한 육종 재료와 관련하여 변이에 따른 종실의 크기, 생육적 특성, 식물체 외형적 변화, 그리고 그 형질들의 유전성 정도에 대한 개념이 필요하다(Chand et al., 2008). 대다수의 보리 품종은 자가 수분 작물이기 때문에 순계 선발을 기반으로 한다. 또한 보리 육종가들은 높은 생산량, 생물적, 비생물적 스트레스 저항성, 맥아 품질 등의 특성을 개선하기 위해 집단육종법, 계통육종법, 집단선발, 반수체 생산, 웅성불임 촉진 순환선발 등 다양한 전통적인 육종 방법을 사용해 왔고 이를 통해 현대의 많은 우량종 개발에 성공했다(Miedaner et al., 2019). 그리고 보리는 잡종 육종도 가능하여 다양한 혼성화 방법을 통해 다수의 잡종 품종이 나오게 되었다(Longin et al., 2012). 하지만 전통적인 방법으로 육종된 품종들은 현재 요구 사항을 충족하지 못할 수 있기 때문에, 분자 육종 도구의 사용이 중요시되고 있다(Eglinton et al., 2006). 이때 보리 분자 육종은 분자 마커 적용, 형질전환, 조직 배양, 이중 반수체 생산과 같은 기존 육종과 생명 공학 방법의 통합을 나타낸다. 보리 육종 프로그램에서 현대 기술과 기존 육종 방법의 통합은 처음 잡종에서 품종 개발까지의 시간을 줄여 육종과정을 가속화시키고 동시에 평균 수확량 증가, 생물적 스트레스, 비생물적 스트레스 저항성과 품질 특성을 개선할 수 있다(Jiang, 2015; Verstegen et al., 2014). 특정 식물을 개선하고자 식물 DNA를 조작하는 분자 육종(Acquaah, 2009)중에서 marker- assisted breeding (MAB)을 이용하여 가치 있는 형질을 가진 식물을 선택하여 고전 식물 육종을 보완하고 더욱 효율적인 육종을 가능하게 했다(Jiang, 2013). 또한 MAB는 marker-assisted selection (MAS), marker-assisted backcrossing (MABC), marker-assisted recurrent selection (MARS) and genome wide selection (GWS) or genomic selection (GS)과 같은 많은 육종 전략을 설명하는데 사용된다(Ribaut et al., 2010). 그리고 분자 마커 기술의 출현으로 대용량 유전형 분석을 통해 보리에서 농업적으로 가치 있는 형질들을 지도화하고 분자표지를 지정할 수 있게 되었고(Verstegen et al., 2014), 게놈 구조 연구를 통하여 목표 유전자를 찾고 식물 육종 과정의 효율성을 증대시켜왔다(Acquaah, 2009). 이와 같은 다양한 접근법은 보리 수확량, 품질 향상, 생물적, 비생물적 스트레스 내성을 포함한 적응 특성의 조합을 가진 품종을 개발하기 위한 주요한 방법들이라고 할 수 있다(Miedaner et al., 2019).
많은 육종 프로그램에서 최우선 순위는 여러 병원체와 해충에 대한 병 저항성을 가진 품종의 개발이다. 보리는 관리요구도가 낮은 작물로 간주되며, 따라서 화학적 방제를 사용하는 것이 경제적으로 좋지 않기 때문에 가장 쉽고 효과적인 방법은 저항성 품종을 재배하는 것이다(Mathre & Mathre, 1997). 보리의 병은 식물체 모든 부분에서 감염될 수 있고 특히 뿌리, 근두썩음은 뿌리 발달과 식물 생존을 심각하게 제한하여 뿌리가 충분한 물을 흡수할 수 없기 때문에 건기 또는 구릉지와 같이 배수가 잘 되어 수분 보유가 어려운 재배지에서 특히 심하게 발생하는 양상을 보인다. 녹, 얼룩, 잎 마름병과 같은 식물의 잎에 발생하는 병들은 광합성 능력을 심각하게 감소시켜 생산량을 감소시키고 종자 크기를 줄어들게 한다. 또한 먹깜부기병과 같은 이삭 병 또한 수확량을 감소시킬 수 있다(Horsley et al., 2009). 이러한 병 저항성 품종을 육종하기 위해 표현형 수준에서 유전형수준으로 선택 단계를 이전할 수 있는 분자 마커의 개발은 원하는 저항성, 생산량, 품질 조합을 목표로, 보다 효율적인 보리 육종을 위한 새로운 기회를 제공한다(Friedt et al., 2003). 한 예로써 보리황화위축 바이러스(Barley Yellow Dwarf Virus- BYDV)에 대한 내성을 목표로, doubled Haploid (DH) 계통과 분자 마커를 사용하여 저항성 유전자 ryd2, ryd3을 재배종 ‘Post’의 염색체 2H에 양적 형질 유전자좌(QTL)와 결합하여 병저항성이 증가한 사례가 있다(Habekuß et al., 2009). 그리고 노란색 모자이크-바이러스(BaMMV, BaYMV, BaYMV- 2, BaMMV-Teik)에 내성이 있는 재배종 Taihoku A 염색체 4H에는 BaMMV 내성 유전자 rym13을 가지고 있으며, 이는 전체 바이러스 복합체에 대한 내성이 있음이 보고되었다. 또한 SSRs (Simple Sequence Repeats)과 AFLPs (Amplified Fragment Length Polymorphisms)을 사용하여 집단 분리 분석을 통해 1 cM 거리에서 BaMMV / BaMMV-Teik 저항성 유전자좌에 가장 가까운 마커가 연결된 지도를 구성했다(Humbroich et al., 2010). 또한 오랫동안 흰가루병은 거의 모든 재배 지역의 보리에 중요한 병원균인데 분자 마커를 식별하기 위해 cDNA-AFLP 접근법을 사용하여 염색체 4H에 위치한 흰가루병 내성 유전자 Mlg의 대립 유전자에 의해 분화되는 근동질유전자 보리 계통을 육성했다(Korell et al., 2008).
보리의 핵심집단(core-collection)은 H. vulgare의 지질학적 기원과 이삭의 row number에 따라 SNP 마커들을 이용하여 33,176계통에서 2,417계통을 확립하였으며 이는 현재 육종 및 유전적 다양성 연구에 집중적으로 활용되고 있다(Munoz-Amatriain et al., 2014, Table 1).
Table 1.
Core collection published for crops belonging to Pooideae subfamily.
| Plant | Markers used | Germ-plasm | Core collection | Reference |
|
Wheat (Triticum aestivum L.) |
Genomic-SSR, EST-SSR | 3942 | 372 | Balfourier et al., 2007 |
| SSR | 23090 | 5029 | Hao et al., 2008 | |
|
Rye (Secale cereal L.) | SSR | 367 | 25 | Targonska et al., 2016 |
|
Barley (Hordeum vulgare) | SNP | 33176 | 2417 | Munoz-Amatriain et al., 2014 |
|
Oat (Avena sativa) | ISSR | 91 | 21 | Boczkowska et al., 2016 |
|
Kentucky bluegrass (Poa pratensis) | RAPD | 362 | 38 | Johnson et al., 2002 |
호밀
호밀(Secale Cereale L.)은 과거 세계의 많은 작물들과 유사하게 근동의 Fertile Crescent 지역에서 처음으로 순화된 작물로 알려져 있으며(Moore et al., 2000), S. cereale은 초기 인류의 정착지인 근동 지역에서 Abu Hureyra I에서 Abu Hureyra II까지 나타났다. S. montanum을 포함한 야생 호밀 종들은 신석기 시대동안 Abu Hureyra 지역 근처에서 발견되었고(Hillman et al., 2000), 순화된 호밀인 S. cereale과 야생 종 S. montanum은 자연상태에서 상호 교배가 가능하다(Zohary et al., 2000).
야생 호밀은 Abu Hureyra 정착 700년 후 Epi-Palaeolithic에서 재배되었고, Abu Hureyra I에서 경작된 밭을 잡초 층상 유적을 발견한 것을 통해 야생 호밀이 순화 되기 전에 먼저 재배되었다(Hillman et al., 2000). 순화된 호밀은 Abu Hureyra I에 이어서 Abu Hureyra II에도 발견됐으나(de Moulins, 2000) 호밀은 근동지역에서 작물로서 재배되지는 않았고 보리와 밀 서식지에서 잡초로 서식한 것으로 보인다(Geiger & Miedaner, 2009). 일년생 호밀 형태는 이러한 보리와 밀의 서식지에서 자연 선택에 의해 탈립성이 적거나 없고, 더 큰 종자, 휴면성이 있는 형태(초기 호밀은 휴면성이 타파되지 않은 것으로 생각됨)로 진화했다(Kranz, 1973). S. cereale은 기원지의 여러 아종들로부터 지금 종으로 진화하는 동안, 약한 포영이 있는 좁은 알곡, 1차 서식지에서의 존재와 S. cereale과의 근친교배가 가능하다는 특징들을 통해 호밀의 야생 조상은 S. vavilovii로 판단되고 있다(Damania et al., 1999; Davies & Hillman, 1992; Frederiksen & Petersen, 1998; Hammer, 1990; Hammer et al., 1987; Hillman et al., 2000; Sencer & Hawkes, 1980; Zohary et al., 2000). 처음 재배된 S. cereale은 기원전 1800년경에 헝가리, 폴란드, 체코, 슬로바키아 공화국에서 정착하면서 시작되었다(Zohary et al., 2012). 기원전 500~400년부터 호밀은 중부 및 동유럽의 많은 지역에서 주요 겨울 작물로서 널리 재배되었다(Behre, 1992).
호밀의 주요 육종 목표는 수확량, 내도복성, 병 저항성이며 추가적으로 제빵용으로 활용하기 위해서는 단백질 함량이 낮으면서 동시에 높은 펜토산 함량을 지닌 호밀을 육종하는 것이 목표이며, 사료용으로는 높은 단백질 함량과 더불어 낮은 펜토산 함량을 지닌 호밀을 육종, 바이오 메탄 생성을 위해서는 높은 건물량이 되도록 육종하는 것이 목표이다(Miedaner et al., 2019). 그 중에서 호밀의 병 저항성은 호밀의 주요 육종 목표로, 호밀의 중요한 질병은 홍색설 부패병(Microdochium nivale), Helgardia herpotrichoides, H. acuformis (syn: Pseudocercosporella herpotrichoides var. herpotrichoides, var. acuformis), M. nivale, Fusarium 종들로 인한 밑둥썩음병, Blumeria graminis f.sp. secalis로 인한 흰가루병, Puccinia recondite로 인한 잎 녹병, P. graminis sp. secalis로 인한 줄기녹병, Rhynchosporium secalis 및 다른 균류들로 인한 잎마름병, Claviceps purpurea로 인한 맥각병, 그리고 M. nivale과 다양한 Fusarium 종들에 의한 Fusarium 이삭마름병(FHB)이 있다. 토양 매개 곰팡이 Polymyxa graminis에 의해 전염된 토양 매개 곡물 모자이크 바이러스(SBCMV), 토양 매개 밀 모자이크 바이러스(SBWMV)와 밀 얼룩 줄무늬 모자이크 바이러스(WSSMV)는 독일의 일부 지역에서 새로운 질병을 유발하였다(Kastirr et al., 2006). 흰가루병과 잎 녹병의 경우 질적, 양적 저항성이 모두 보고된 반면, 밑둥썩음병, 이삭 마름병, 맥각병에 대한 양적변이가 발견되었다(Geiger & Miedaner, 2009). 호밀은 일반적으로 내병성 및 내한성이 우수한 작물로 알려져 있지만 일부 생산에 영향을 미치는 병들에 대한 저항성 육종이 필요하다. 밀이나 보리와 같은 자가수정 작물과 비교할 때, 타가수정 작물인 호밀의 개체군은 자연선택에 의해 축적된 저항성을 더 많이 보유할 가능성이 높다(Geiger, 1988; Mirdita, 2006). 또한 근계를 유지하기 위해 half- 혹은 full-sib family를 기반으로 순환선발방법을 통해 질병 저항성을 개선할 수 있다(Geiger & Miedaner, 2009). 유전자형 분산은 개체군에서 함께 분리되는 양적, 질적 저항성들에 의해 야기되는 흰가루병, 잎 녹병 저항성에 대해 매우 큰 것으로 밝혀졌다(Wilde et al., 2006). 이 경우, 모든 양적 저항성에 대해 치명적인 병원체 종을 사용하여 종 특이적 저항성 유전자의 차폐 효과가 제거된 경우에만, 양적 저항성이 발견될 수 있다. 밑둥썩음병 저항성의 경우, 유전자형 분산이 중요하지만, 일반적으로 자가임성 개체에서는 낮다(Miedaner et al., 1995). 또한, Fusarium 이삭마름병 저항성의 경우, 자식성 계통 간의 유전자형 분산이 크기 때문에 쉽게 이용할 수 있고, 맥각병 저항성에 대한 분산은 세포질적-유전자적 웅성불임 자가불화합성 개체군을 포함하여 지금까지 테스트된 모든 개체에서 사용할 수 있다(Mirdita, 2006). 잡종 육종에서 흰곰팡이와 잎 녹병 저항성은 이미 초기 자가생식 세대를 선별하고, 다른 모든 병의 경우에는 우량 개체에서 대립 유전자 빈도 증가를 목표로 하는 특정 육종 전 절차가 필요하다. 밑둥썩음병과 맥각병 저항성을 향상시키기 위해 자가불화합성 우량종 개체군 혹은 유전 자원에서 양성 대립 유전자 이입을 통해 유전적 분산을 증가시키는 것이 좋다(Geiger & Miedaner, 2009).
호밀의 핵심집단은 Secale cereal L.의 367 계통을 SSR 마커를 이용하여 역사적으로나 현대의 품종, 경작 재료, 재래종 및 육종품종에 따라 지질학적 다양성에 따른 25계통의 핵심집단을 형성하였다(Targonska et al., 2016). 호밀의 유전적 다양성과 관련된 연구는 더 심도 있게 진행되어야 할 필요가 있을 것으로 생각된다.
귀리
귀리(Avena sativa) 기원의 중심은 알려지지 않았으나 다른 Pooideae 식물들과 같이 지중해 분지 또는 중동 지역에 있을 가능성이 높다. 귀리의 순화는 밀과 보리보다 훨씬 늦게 발생했으며 호밀과 비슷한 양상으로서 밀과 보리 경작지에 존재하는 잡초 귀리에서 선택되었을 것으로 추정된다(Murphy & Hoffman, 1992). 귀리는 밀과 같이 기본염색체수가 7인 2배체, 4배체, 6배체등의 다양한 배수성이 발견된다. 처음 재배된 귀리는 3개의 2배체 게놈(AA, CC, DD)의 집합체에서 유래한 2n=6x=42의 6배체다. 6배체의 D 게놈은 A 게놈과 매우 유사하며 아마도 최근 변종일 것으로 예상되며, 현존하는 DD 게놈 이배체는 확인되지 않았다(Leggett & Thomas, 1995). 분류자에 따라 15개 혹은 16개의 A게놈 이배체 종들은 그들 사이에 만들어진 F1 하이브리드의 상호임성에 따라 As, Al, Ac 하위 그룹으로 나누기도 한다. C 게놈 2 배체는 A.clauda와 A.eriantha의 두 종으로 구성된 Cp와 A. ventricosa 한 종으로 구성된 Cv 2개의 하위 그룹이 있는 3종의 그룹이다. C 게놈 그룹의 구성원은 A 게놈 그룹에서 유전적으로 분리되며 세포질 리불로스 2인산 카르복실라제 형태(Steer, 1975), 엽록체 DNA (Murai & Tsunewaki, 1987), 높은 이질염색체 구성(Fominaya et al., 1988) 등으로 구별된다.
4배체 귀리 종에는 세 가지 유형이 있다. 첫 번째, AABB에서 B 게놈 염색체는 A 게놈 염색체와 매우 유사하여 동질배수체 기원을 암시한다. 그러나 이 종은 이염색체 유전성을 보여주며(Leggett, 1992) 위성 DNA 서열이 두 세트의 구성 염색체를 구별하는 것으로 보고되었다(Irigoyen et al., 2001). AABB 종에는 반 온대 지역에서 흔히 볼 수 있는 잡초종인 A. barbata와 에티오피아 고원의 A.vaviloviana에서 순화된 A.abyssinica가 포함된다(Ladizinsky, 1989). AACCDD 6배체의 조상일 가능성이 있는 AACC 그룹은 지중해 서부 지역의 A. maroccana (magna)와 A. murphyi, 그리고 최근에 발견된 A. insularis가 있는데, A. insularis는 6배체 귀리와 F1 잡종에서 더 높은 interspecific chromosome pairing frequency 때문에 6배체 귀리의 실제 조상 종에 더 가까운 것으로 생각된다(Ladizinsky, 1998). 세 번째 유형의 귀리 4배체인 A. machrostachya는 유일한 다년생 종이며, 그것은 또한 자연적으로 교잡수정, 이질4배체이며 AA, CC 게놈 이배체와 먼 관계만을 보여준다(Leggett & Thomas, 1995).
주요 6배체 종은 추파성 잡초 종 A. sterilis, 춘파성 잡초 종 A. fatua 재배 종 A. sativa를 포함한다. 남서부 유럽에서 시작된 추파성 재배 귀리는 일부 분류 학자에 의해 별도의 붉은 귀리 종인 A. byzantina로 분류되었다(Rines et al., 2006). 과거에는 모든 6배체 귀리가 높은 교잡성 때문에 단일 종인 A.sativa로 간주되어야 한다고 제안했지만(Ladizinsky & Zohary, 1971) 귀리 연구자들은 종자 분리 차이를 기반으로 6배체 귀리의 재배종과 잡초 종 분리를 유지했다. 하지만 껍질이 없는 귀리 영과의 쉬운 탈곡성으로 인해 처음에는 별도의 종인 A.nuda L.가 분류되었지만, 겉껍질이 없는 형질이 주로 몇 가지 변형을 가진 단일 유전자에 의해 지배된다는 것을 통해 6배체의 겉껍질이 없는 귀리는 현재 A.sativa의 변종으로만 간주된다(Rines et al., 2006).
귀리 육종 중 귀리 병 저항성은 귀리 유전형질 개선의 주요 목적이다. 일반적인 귀리 질병에는 관녹병, 줄기녹병, 흰가루병, 겉깜부기병, 속깜부기병, 푸사리움적매병, spetoria 잎마름병이 있다(Campbell et al., 2000; Haber & Harder, 1992). 귀리 병 저항성을 위한 몇 가지 육종법은 주요 종 특이적 유전자의 통합, 부분 저항성 선택, 유전자 피라미딩, 여러 계통의 품종 또는 변종 사용 등이 있다(Brown, 2015; Cabral, 2009; Cabral et al., 2014; Carson, 2009; Jackson et al., 2010; Long et al., 2006; Rubiales & Niks, 2000). 그 중에서 부분 저항성(비 종 특이적 또는 성체 식물 저항성)은 감염 발생을 완전히 예방하지는 않지만 잎당 농포 수, 농포 크기, 포자 생산을 줄이고 농포 발생 잠복기를 증가시킨다는 많은 연구가 있다. 따라서 부분적 저항성은 높은 수준의 저항성을 부여하지는 않지만 병원균 발생에 대한 지연 효과로 인해 병의 전염을 예방하고 장기적인 해결책을 제공할 수 있다(Long et al., 2006; Portyanko et al., 2005; Zambonato et al., 2012). 또한 저항성 유전자형에 대한 스크리닝과 이를 교잡 프로그램에 포함시키는 것도 포함된다. 대부분의 경우 지속적인 저항성은 소수 종 비특이적 유전자의 복합체 또는 소수 유전자와 주요 종 특정 유전자의 조합에 기반한다. 주요 유전자와 소수 종 비특이적 유전자를 포함하는 식물을 표현형별로 선택하거나, 주요 유전자가 2개, 3개 이상인 유전자형(피라미드 유전자)에서 1개의 주요 유전자가 있는 유전자형을 분리하는 것은 불가능할 수 있다. 따라서 marker assisted selection (MAS)은 하나의 유전자형에서 여러 저항성 유전자의 검출을 가능하게 하여 유전자의 피라미드화를 단순화 시킬 수 있기 때문에 가장 광범위하게 육종에 활용되고 있다(Gorash et al., 2017).
관녹병(Puccinia coronata f.sp. avenae에 의해 유발됨)은 전 세계적으로 가장 널리 퍼져 있고 중요한 귀리 질병으로, 귀리가 재배되는 모든 환경에서 발생한다(Gnanesh et al., 2014; Graichen et al., 2011; Leonard & Martinelli, 2005; Montilla- Bascón et al., 2015; Sebesta et al., 2003; Sikharulidze et al., 2015). 미국에서는 1919년(Roelfs & Bushnell, 1985)에 관녹병 저항성 육종을 시작했으며 현재까지 100개 이상의 내성 유전자가 밝혀졌다(Cabral et al., 2014; Gnanesh et al., 2014; Gnanesh et al., 2015). 1960 년대 이후 육종가들은 주로 야생 6배체 귀리 A. sterilis의 일련의 주요 저항성 유전자를 통합하여 저항성 형질을 획득하였다. PCR 기반 마커 유전자좌인 Pc68 관녹병 저항성 유전자좌는 원래 연결된 random amplified polymorphic DNA (RAPD) 마커 UBC269를 사용하여 규명되었으며(Penner et al., 1993). 과거 유전 분석에서 Pc68 유전자좌가 Pg3, Pg9 줄기녹병 저항성 유전자좌뿐만 아니라 관녹병 저항성 유전자좌와 클러스터링되었음을 보여주었고, Pg3는 동일한 UBC195 RAPD 마커뿐만 아니라 UBC458 RAPD 마커와 여러 RFLP와 저장 단백질 마커에 의해 RAPD 마커 ACOpR-2 (acor195 또는 UBC195라고도 함), Pg9로 규명되었다(O'Donoughue et al., 1996). 그러나, 이들 Pc 종 특이적 유전자 각각의 저항성은 이 유전자를 포함하는 품종 육종 후 관녹병 독성 패턴의 급격한 변화로 무너졌다. Pc38과 Pc39를 포함한 Pc 유전자의 조합을 사용한 후 Pc68을 추가하면 효과 기간이 연장되는 것을 보였지만(Chong & Zegeye, 2004), 그 효과는 거의 사라졌다. 또한 MAS 목적으로, 녹병 저항성 유전자 Pc94를 피라미드화하기 위해 Pc94 (Chong et al., 2004)용으로 두 개의 Sequence Characterized Amplified Region (SCAR)이 개발되었고, 추가적으로 SCAR는 Pc38 유전자좌에 연결되었다(Wight et al., 2005). 귀리의 관녹병에 대한 부분적인 저항성은 MN841801-1 계통(Portyanko et al., 2005), 귀리 품종 URS 21 (Zambonato et al., 2012), UFRGS 계통(Chaves et al., 2004)과 같은 여러 유전자형에서 확인되었다. 그리고 MN841801-1 계통에서 성체 식물 저항성(APR)과 관련된 분자 마커를 조사하여 4개의 주요 양적 형질 유전자좌(QTL)와 3개의 작은 QTL을 검출했다(Portyanko et al., 2005). Acevedo et al. (2010)은 동일한 귀리 계통에서 부분 저항성과 관련된 추가 QTL을 발견했으며, Lin et al. (2014)는 MN841801에서 표현형 APR 발현의 최대 76%를 설명하는 추가적인 주요 QTL을 발견했다.
Puccinia graminis sp. avenae로 인한 귀리 줄기녹병은 여러 귀리 재배 지역에서 심각한 수확량의 손실을 유발할 수 있다. 캐나다의 귀리는 Pg2와 Pg13 유전자 조합을 통해 수년 동안 효과적인 줄기녹병 저항성을 가지고 있다. 그러나 이러한 Pg 유전자에 독성을 갖고 있는 줄기녹병 종 NA67, NA74가 발생했다(McCallum et al., 2002). 이러한 녹병에 대한 효과적인 내성은 이배체 귀리 A. strigosa에서 확인되었다(Fetch & Dunsmore, 2004). 그리고 최근에는 17개의 귀리 줄기녹병 저항성 유전자(Pg 유전자)와 Pg-a 복합체가 설명되었다(Gnanesh et al., 2014).
흰가루병(Blumeria graminis sp. Avenae)은 유럽의 서늘하고 습한 지역과 동유럽 일부 국가에서 가장 중요한 귀리 엽면병이다(Okon, 2015; Roderick et al., 2000). 귀리의 흰가루병에 대한 내성은 귀리 곰팡이 내성(Oat Mildew Resistance, OMR) 그룹으로 특성화된 주요 유전자에 의해 좌우되며(Jones & Jones, 1978), 지금까지 6개의 OMR 그룹과 흰가루병에 대한 7개의 저항성 유전자가 설명되었지만(Hsam et al., 2014; Lai et al., 2006; Okon, 2012; Okoń, 2015; Roderick et al., 2000; Yu & Herrmann, 2006) 3개(OMR1, OMR2, OMR3)는 육종 프로그램에서 일반적으로 사용되었다(Hsam et al., 2014; Okon, 2012; Okoń, 2015).
귀리의 핵심집단은 Avena sativa의 agro-morphological 데이터인 The mean difference (MD%), The variance difference (VD%), The variable rate of the coefficient of variance (VR%), The coincidence rate of range (CR%)에 따라 ISSR마커(Inter-Simple Sequence Repeats)를 이용해 91계통에서 21계통의 핵심집단을 형성하였다(Boczkowska et al., 2016). 이는 실제 활용이 어려운 수준이기 때문에 다양한 수집계통을 통한 핵심집단의 확장이 필요하다.
한지형잔디
Pooideae아과에서 한지형 잔디로 분류될 수 있는 종은 대부분 Agrostis, Festuca, Lolium, Poa 속에 속하는 식물들로서 목초로 분류가 되고 있다(Catalán et al., 2004; Davis & Soreng, 2007; Macfarlane & Watson, 1980, 1982; Quintanar et al., 2007; Saarela et al., 2010; Schneider et al., 2009; Schneider et al., 2012; Soreng et al., 1990; Soreng et al., 2007). 한지형 잔디는 경제적 가치로 인해 많은 관심을 갖게 되었고, 다양한 육종 프로그램을 통하여 스트레스 내성이 증가되고, 유전적 개선을 통해 해충 저항성이 향상된 잔디 품종들이 나오게 되었다(Meyer & Funk, 1989). 잔디 개량의 주요 발전은 내생 진균을 다년생 라이그래스, 톨페스큐, 파인페스크류의 잔디 유형 품종에 혼입한 것이다(Funk et al., 1985; Funk et al., 1983; Funk et al., 2018; Murphy et al., 1993; Saha et al., 1987).
켄터키블루그래스(Poa pratensis)는 가장 널리 재배되는 한지형 잔디 중 하나로, 넓은 뿌리 시스템을 통해 우수한 잔디 강도와 스트레스 후 빠른 회복력을 갖고있다(Bonos et al., 2006a). 하지만 Drechslera poae에 의한 잎 반점, Puccinia graminis에 의한 줄기녹병, Ustilago striiformis에 의한 줄무늬깜부기병, Sclerotinia homoeocarpa에 의한 줄녹병 같은 곰팡이 병원균에 의해 피해를 받을 수 있다. 그리고 켄터키블루그래스는 무수정생식을 통해 번식을 하는데, 무수정생식을 이용하면 다양한 환경에 적응하고 향상된 병 저항성 잔디 육종을 통해 고유의 유전자형 개발이 가능하다(Funk, 2000). 그래서 1960년대 후반 CR Funk와 New Jersey Agricultural Experiment Station (NJAES)은 종간 및 종내 교잡을 통해 고도로 무수정생식을 하는 식물 개발 잡종형성기술을 완성했다(Bailey et al., 1995; Bonos et al., 2005a; Bonos et al., 2004; Funk & Sang, 1967; Meyer et al., 1984; Pepin & Funk, 1971). 또한 켄터키블루그래스 잎 반점 저항성을 높이기 위해 Merion켄터키블루그래스, Midnight켄터키블루그래스 같은 품종들이 개발되었고(Funk et al., 1970; Halisky & Funk, 1966; Halisky et al., 1966; Morris, 1996a; Morris, 1996b), 이러한 켄터키블루그래스 생식질을 선별하기 위한 스크리닝 기술을 개발하였다(Czembor, 2002; Czembor, 2003). Funk et al.과 NJAES의 다른 육종가들은 켄터키 블루그래스 생식질을 선별하여 잎 반점에 대한 내성을 찾기 위해 필드조건에서 이 병의 자연발생을 활용했다. 또한 임성 재조합을 촉진하기 위한 최적의 조건에서 종내 교잡 기술도 켄터키블루그래스의 잎 반점 저항성을 개선하는데 유용하였다(Pepin & Funk, 1971). 그리고 켄터키블루그래스에 광범위한 손실을 일으키는 다른 병 중 하나인 줄기녹병의 저항성을 높이기 위해 또한 종내 교잡을 통해서 새로운 잡종을 개발하고 있고(Shortell et al., 2004), 텍사스 블루그래스(Poa arachnifera)와 켄터키블루그래스를 교배하여 생성된 종간 잡종에서 줄기녹병 저항성이 더욱 좋아진 새로운 품종 육종도 하고 있다(Shortell et al., 2004).
톨페스큐는 미국 북부 절반, 캘리포니아 일부 지역에서 널리 사용된 잔디이며 ‘Alta’ (Cowan, 1956)와 ‘Kentucky’ (Fergus & Buckner, 1972)는 사료 목적으로 개발된 품종이다. 톨페스큐에서 가장 흔하고 위험한 병은 갈색잎마름병, 줄기녹병, Drechslera dictyoides에 의해 생기는 망반병, Pyricularia grisea에 의해 유발되는 잔디도열병 등이 있다(Linscombe et al., 1983; Tredway et al., 2003). 육종가들은 톨페스큐의 줄기녹병 저항성 생식질 생산을 위해 톨페스큐 스크리닝 기술을 개발하였고(Welty & Barker, 1993), 이를 통해 많은 우수한 줄기녹병 저항성 품종들을 개발하였다(Hurley et al., 1995; Rose-Fricker et al., 1999; Welty & Barker, 1993).
다년생 라이그래스는 겨울에 온화하고, 여름에는 시원하고 다습한 해양기후에 잘 적응한다(Meyer & Funk, 1989). 과거에 ‘Manhattan’과 ‘Pennfine’ 품종이 나오면서 다년생 라이그래스 사용이 증가했고 종자 생산 분야에서 다년생 라이그래스에 Puccinia graminis subsp. Graminicola에 의해 유발되는 줄기녹병과 회색 잎 반점 같은 질병 발생이 동시에 증가했다. 다년생 라이그래스에서 흔히 발생하는 다른 질병으로는 Drechslera siccans에 의한 갈색잎마름병, 돌반점, Puccinia coronata에 의해 생기는 관녹병, 회색홍색설부패병 등이 있다(Bonos et al., 2006b). 그 중 회색 잎 반점 저항성 육종을 위해 지난 몇 년간 자연 질병 발생과 표현형 및 유전형 순환선발을 통해 회색 잎 반점에 저항성 다년생 라이그래스 35개 계통을 선발하였고(Shortell et al., 2004), 높은 유전성 추정치와 환경적 영향의 결여를 통해 다년생 라이그래스의 회색 잎 반점 저항성이 몇 가지 주요 유전자에 의해 제어된다는 것을 확인하였다. 또한 상가적 유전변이도 저항성을 예측하는데 중요한 것으로 밝혀졌다(Bonos et al., 2005b; Han et al., 2006). 그리고 일년생 라이그래스와 다년생 라이그래스 교배를 통해 회색 잎 반점 저항성 QTL 마커도 확인하였다(Curley et al., 2005). 그리고 다년생 라이그래스의 줄기녹병 저항성 육종을 위해 RoseFricker는 P. graminis subsp graminicola에 감염된 6개의 다년생 라이그래스 교배에서 질병이 더 느리게 진행되고 초월분리가 발생하는 식물을 관찰하여, 양적 저항성이 여러 유전자들과 관련 있음을 보여주었다. 또한 QTL 마커를 통해 다년생 라이그래스에서 관녹병 저항성을 확인하였고(Thorogood et al., 2001), 환경 조건에 따라 다른 것을 밝혔고, 이렇게 확인된 QTL 마커가 귀리의 관녹병 저항성 유전자 클러스터와 syntenic을 이루고 있는 것을 통해 pooideae에서 여러 종에 걸쳐 유전자가 보존되고 있다는 것도 확인하였다(Dumsday et al., 2003). 이를 통해 향후 비교 매핑 분석과 기능적 게놈 연구는 잔디의 녹병 저항성에 대한 이해를 향상시킬 것이다(Bonos et al., 2006b).
켄터키블루그래스의 핵심집단은 현재 미국 농업연구청(USDA-ARS)에서 수집한 전 세계의 Kentucky bluegrass 362계통중 RAPD (Random Amplification of Polymorphic DNA)와 유전자원 및 작물의 진화 형태 데이터를 기반으로 전체 계통에 존재하는 유전적 다양성의 대부분을 포괄하는 38개의 핵심집단을 구성했다(Johnson et al., 2002).
미래연구방향
포아풀아과의 중요한 진화적 특징 중 하나인 내한성의 진화과정에 대한 연구는 현재까지 포아풀아과의 계통 발생에 대한 불확실성으로 인해 신생대 전체에 걸쳐 기온이 내려가는 것이 포아풀아과의 진화 역사에 어떻게 영향을 미쳤는지 아직 정확하게 밝혀지지 않았다. 따라서, 포아풀아과 종들의 비교 전사체 분석 같은 비교 유전체학적 연구와 RNA 미세배열(Micro-array)와 RNA 염기서열분석 등을 통한 내한성과 관련된 유전체 서열을 전사체 정보와 결합한 기능 유전체학적 연구가 필요하다. 또한, 내동성의 중요 기작인 저온 순화와 춘화과정의 유전이 포아풀아과와 관련이 먼 모델 종에서 설명되었기 때문에, 포아풀아과 종 내에서 저온 순화와 춘화에 관련된 유전자 서열 및 각 유전자들끼리의 상호작용을 통한 유전자 복합체의 정확한 메커니즘을 파악해야 할 것이다.
결 론
유전체학과 계통분류학이 발전하면서 육종에 이를 적용하기 위한 많은 연구들이 진행되고 있다. 특히 농경학적으로 가치가 높은 밀, 보리, 귀리, 호밀, 한지형 잔디가 존재하는 화본과의 포아풀아과(Pooideae)에 대한 비교 유전체 분석을 통한 계통학적 연구와 함께, 각 식물들의 기능 유전체 분석을 통해 이를 농업적으로 육종에 활용하여 다양한 유전자원을 개발하고 있다.
우리는 주요 진화 요인 중 하나인 전체 유전체 중복(Whole genome duplication)이 다양한 모델 식물의 유전체 분석을 통해 계속 밝혀지고 있고, 진화적으로 매우 가치가 있는 화본과 식물들의 현재까지 진행된 분류학적 연구들과 함께 화본과에서 가장 많은 종을 보유하고 있으며, 온대 및 한대지역에 넓게 분포하고 있는 포아풀아과의 기원과 진화에 대해서 정리하였다. 또한, 포아풀아과의 비교유전체 연구들 중 기후변화와 서식지 이동에 초점을 맞춘 한대지역 적응성과 관련된 진화 과정에 대해서도 작성하였다. 그리고 포아풀아과 중 맥류 및 한지형 목초를 중심으로 지질학적, 지층학적 연구로 밝혀진 인류의 농업 발전과 더불어 식물 별 순화 과정을 파악하였으며, 전통육종 방법과 과학 발전의 조화를 통해 식물별, 특정 형질별로 다양하고 효율성이 높은 식물 육종 방법들이 개발되었는데, 그 중에서 병 저항성을 목적으로 수행된 연구들을 요약하였다.
이처럼 농업적으로 굉장히 중요한 가치를 지닌 포아풀아과의 많은 관심과 유전 및 육종 연구들이 이루어지고 있지만, 화석 자료의 부재와 화본과 식물이 다른 피자식물보다 느린 염기서열 변화 속도 등으로 인해 아직까지 정확한 포아풀아과의 기원과 진화계통도(Phylogenetic tree) 조차 확실하게 밝혀져 있지 않다. 또한, 포아풀아과의 중요한 특징 중 하나인 내한성의 진화과정은 식물 종의 다양성, 저온 반응성 유전자 기원의 진화론적 연구 부족과 모호성 등으로 인해 아직 정확히 밝혀지지 않았다. 현재 이러한 한계들을 극복하기 위해 신생대의 기후변화와 포아풀아과의 분화와의 연관성에 대한 연구들과 비교 전사체 분석 같은 비교 유전체학을 비롯한 다양한 유전체 기술을 통해 더욱 폭넓은 연구들이 활발히 진행 중이며, 이러한 결과들을 육종에 활용하기 위한 많은 노력이 이루어지고 있다.




