서 론
재료 및 방법
식물재료
콩 역병 저항성 검정
DNA 추출 및 SNP 유전자형 분석
SNP 유전자형에 대한 일원분산분석(single-factor ANOVA)
유전자지도 작성 및 Linkage analysis
결과 및 고찰
대풍 × 천알 재조합자식 계통 집단의 표현형 분리
SNP 유전자형에 대한 일원분산분석(single-factor ANOVA)
대풍 × 천알 집단의 유전자 지도 작성
유전자지도를 이용한 linkage analysis
천알의 역병 저항성 유전자좌 발견의 의의
적 요
서 론
콩(Glycine max L. Merr.)은 우리나라를 포함하는 동아시아 지역을 원산지로 하는 작물로 특히 우리나라에는 콩을 활용한 다양한 식품이 소비되고 있다. 2022년 기준 국내에서는 63,956 ha에서 129,925 ton의 콩이 생산되었다(Statistics Korea, 2022). 기후 변화와 더불어 발생하는 병해충의 발생은 세계적으로 콩 생산량을 저해하는 주된 요인 중 하나로, 불마름병, 역병, 모자이크바이러스, 진딧물 등이 보고되고 있다.
콩 역병은 토양 유래 난균(oomycete)인 Phytophthora sojae Kaufmann and Gerdemann에 의해 발생하는 병해로, 콩의 주산지인 북미 지역과 캐나다를 중심으로 1996년부터 2014년 사이 매년 평균 100만 톤에 해당하는 수량 손실을 야기하는 것으로 추산되었다(Allen et al., 2017; Koenning & Wrather, 2010; Wrather et al., 2001; Wrather & Koenning, 2006; Wrather & Koenning, 2009). P. sojae는 주화성(chemotaxis)을 가지고 있어 콩의 뿌리에서 분비되는 이소플라본을 인지하고 이동하여 감수성 식물체의 뿌리를 감염시키는 것으로 알려져 있다(Morris & Ward, 1992). 유주자낭이 물속에서 유주자를 퍼트리고 뿌리에 닿은 유주자는 관다발 조직에 침투하게 되어 유주자가 식물 뿌리의 감염시키는 방식이다(Morris & Ward, 1992). 따라서, 콩 역병은 우리나라 논토양과 같이 습하거나 배수가 불량한 토양 환경에서 토양 수분이 포화 상태가 되면 그 발생의 정도가 증가한다. 감수성 식물체가 유묘기에 감염되면 바로 시들고(damping-off), 성체기에는 뿌리가 썩기 시작하고 점차 지상부(줄기)가 썩어 식물체가 고사하게 된다(Dorrance & Schmitthenner, 2000). 콩 역병은 유묘기부터 생장 후기까지 전체 생장 기간에 걸쳐 감수성 식물체에서 발생할 수 있고 역병균은 토양 내 또는 식물체 파편에서 수년 간 월동과 생존이 가능하기 때문에 화학적 방제로는 한계가 있다.
이런 이유로 역병의 피해를 효과적으로 줄일 수 있는 지속 가능하고 친환경적인 전략은 역병 저항성 품종을 이용하는 것이다. 감수성인 우량 품종에 저항성 유전자(R-gene)를 도입하거나 복수의 저항성 유전자를 집적(gene stacking)함으로써 신품종을 육성하는 것이 안정적인 생산 도모와 환경 친화적 측면에서 가장 각광받는 방법이다. Rps (Resistance to Phytophthora sojae)로 명명된 역병의 저항성 유전자(R-gene)는 여러 품종 및 유전자원으로부터 보고되었다. 지금까지 10개 염색체에서 30개 이상의 역병 저항성 유전자 또는 대립유전자(allele)들이 보고되었고, 다수의 연구에서 염색체 3번, 13번, 18번에 위치한 유전자좌를 반복적으로 보고하였다(Burnham et al., 2003; Cheng et al., 2017; Demirbas et al., 2001; Jang et al., 2020a, 2020b; Li et al., 2017; Lin et al., 2013; Niu et al., 2017; Sahoo et al., 2017; Sandhu et al., 2004; Sugimoto et al., 2011; Sugimoto et al., 2007; Sun et al., 2014; Sun et al., 2011; Weng et al., 2001; Yu et al., 2010; Zhong et al., 2017). 식물-병원균 상호작용에서 R 유전자로부터 생성되는 R 단백질은 병원균에서 분비되는 비병원성(avirulence, Avr) 단백질을 인식해 저항성 또는 감수성을 보인다(Flor, 1951). 동일한 병원균의 각 균계(race)에 따라 Avr 단백질은 다른 구조를 가지며, R 단백질과의 상호작용은 균계에 따라 특이적인 특성을 가진다.
콩의 표준 유전체 정보를 활용한 분자 마커 기술은 매우 고도화되었다. 대용량 염기서열 분석이 용이해짐에 따라 대량의 SNP 마커를 확보할 수 있게 되고, 대량 SNP 칩 또는 genotyping by sequencing (GBS)를 이용한 고속 대량 SNP genotyping과 고밀도 유전자지도 작성이 가능해졌다. 또한, 대량 유전자원에 대한 표현형 결과를 이용해 전장유전체 연관 분석(Genome-wide association study, GWAS) 및 전사체 분석이 활발하게 이루어지고 있다(Cai et al., 2019; Van et al., 2021). 최근 역병 저항성 유전 분석 연구에서는 F2 집단을 재료로 한 linkage mapping을 통해 염색체 3번 5.8Mb 영역에 Rps14를 보고하였고(Chen et al., 2021), 고밀도 유전자지도 작성을 통해 기존에 밝혀졌던 Rps 영역을 정밀하게 좁히거나(Jiang et al., 2020; Zhong et al., 2020), 대량의 유전자원에 대한 GWAS분석을 통해 복수의 역병 저항성 유전자좌 연관 SNP들을 보고하였다(Guo et al., 2022; Li et al., 2022; Rolling et al., 2020; Van et al., 2021; Zhao et al., 2020).
우리나라에서는 충남 홍성과 청양 지역에서 처음 콩 역병이 발견되어 균이 분리되었으나(Jee et al., 1998), 그간 연구가 거의 없다가 최근에 역병 저항성 육종 연구가 활발하다. 국내 역병 균주 4개에 대해서 국내 콩 품종 20개에 대한 역병 저항성 여부를 보고했다(Kang et al., 2019). 연구에 이용된 품종들은 하나 이상의 역병 균주에 대해 감수성을 보였고, 대풍을 포함한 10개 품종은 이용된 균주 4개에 대해 모두 감수성을 나타냈다. 최근에 2개의 품종(‘대원’, ‘소청2호’)에서 역병 저항성 유전자좌가 밝혀졌고(Jang et al., 2020a, 2020b), 추가로 다른 품종 및 재배콩 핵심집단에서 타 역병 균주에 저항성 유전 분석이 진행 중이다(unpublished). 본 연구에서는 ‘대풍’ × ‘천알’ 재조합 자식 계통(recombinant inbred line, RIL) 집단을 이용하여 역병 균주 ‘KACC.40468’ (이하 균주40468로 표기)(Kang et al., 2019)에 대한 저항성 유전자좌를 탐색하고자 표현형 검정 및 linkage analysis을 수행하였다.
재료 및 방법
식물재료
본 연구에서는 대풍 × 천알 재조합자식 계통 집단을 이용해 천알의 역병 저항성 유전자의 위치를 탐색하고자 했다. 대풍은 내도복성과 고수량성을 가지는 장류용 품종이고(Park et al., 2005), 천알(계통명 SS0404-T5-76)은 ‘풍산나물’ × ‘SS2-2’ 교배집단에서 선발된 우량 계통으로 다분지 고수량 계통이다(Shim et al., 2017). 선행 연구에서 대풍은 콩 역병 균주40468에 대해 감수성을 보였고(Kang et al., 2019), 예비 실험에서 천알은 저항성을 보였다(Fig. 1). 대풍과 천알은 2012년에 단국대학교에서 처음 인공교배를 실시하여, 동년 가을 5개 F1 종자를 얻었다. 그로부터 세대 진전을 거쳐 56개 F6 계통으로 이루어진 집단이 만들어졌다. 2019년에 종자를 분양 받아, 충남대학교 실험 농장(대전)에 파종하여 종자를 증식하였고, 이 중 이용가능한 38개 계통이 표현형 검정에 이용하였다.
콩 역병 저항성 검정
유전분석을 위해 대풍 × 천알 재조합자식 계통 집단에 대해 콩 역병 균주40468 에 대한 저항성 검정을 수행했다. 전제 집단 중에서 가용한 종자 수가 충분한 38개의 계통만 검정에 이용하였다. 저항성 검정은 하배축 접종(hypocotyl inoculation) (Dorrance et al., 2004) 방법으로 수행했다. 접종원은 V8 배지에 역병 균주를 치상한, 배지들을 25°C 암실에서 약 10일 동안 항온 배양했다. 동시에 각 계통 별 10~15립의 종자를 원예상토를 채운 플라스틱 포트(지름 13 cm, 높이 10 cm)에 파종한 후, 식물 생장상(25°C, 낮:밤 14:10시간)에서 발아시켰다. 파종 7일차에, 하배축 상단의 중심부를 의료용 칼날로 세로 방향 약 1 cm 길이로 절개하였다. 역병균으로 덮인 V8 배지는 50 mL 주사기에 2회 통과시키는 방법으로 잘게 부쉈고, 18게이지(G) 바늘이 장착된 10 mL 주사기를 이용해 식물 개체 당 0.2-0.4 mL씩 절개 부위에 충전하는 방식으로 접종했다. 접종된 식물체는 24 시간 동안 >90% 습실 처리 후, 식물 생장상에 6일간 두었다(25°C, >80%, 낮:밤 14:10 시간 주기). 접종 7일차에 각 계통 별로 전체 유묘 개수에 대한 고사 유묘 개수의 비율을 얻었다. 이때 고사 유묘 비율이 20% 이하는 저항성, 80% 이상은 감수성, 20% 초과 및 80% 미만인 경우에는 중간형(intermediate)으로 결정했다. 모본과 부본은 매회 반복적으로 평가되었고, 각 계통에 대한 검정은 3회 반복했다.
DNA 추출 및 SNP 유전자형 분석
대풍, 천알, 각 계통의 식물체로부터 어린잎을 채취하여 동결 건조한 후 갈아서, cetyl trimethyl ammonium bromide (CTAB) 방법(Doyle & Doyle, 1987)으로 DNA를 추출했다. Axiom 180K SoyaSNP array (Affymetrix, Santa Clara, USA)를 이용하여 각 계통에 대해 SNP 유전자형 분석을 수행했고, 38개의 계통에 대해 169,028개의 SNP 유전자형 데이터를 얻었다. 먼저, 이 데이터에서 모본과 부본 간 다형성이 없거나 불확실한 유전자형을 보이는 SNP 146,357개를 제거하여 22,671개 SNP으로 추렸다. 이어서 결측 값과 이형 접합형(heterozygosity)의 비율이 전체의 20% 이상인 SNP 34개를 제거하고 22,637개의 SNP이 남았다. 결과적으로, 38 계통에 대한 22,637개의 SNP 유전자형 데이터를 가지고 이후 유전분석을 수행했다.
SNP 유전자형에 대한 일원분산분석(single-factor ANOVA)
대풍 × 천알 집단의 재조합자식 계통의 균주40468에 대한 병 반응과 통계적으로 연관된 SNP을 찾기 위해 분산분석(analysis of variance, ANOVA)을 수행했다. 계통들을 22,637개 SNP의 대립유전자형(allele)에 따라 두 개 그룹으로 분리한 후 각 그룹에서 나타난 병 반응(유묘의 고사율)의 평균값의 유의성 여부를 통계분석프로그램 R (Team, 2019)의 패키지 lme4 (Bates et al., 2015)를 이용해 분석했다.
유전자지도 작성 및 Linkage analysis
대풍 × 천알 재조합자식 계통 집단의 유전자지도를 작성하고자 유전분석 프로그램인 IciMapping version 4.2를 이용했다(Meng et al., 2015). SNP 전처리로부터 얻은 22,637개의 SNP으로부터 BIN 기능을 이용해 중복성이 높은 SNP을 제외했고, 그 결과 2,513개의 SNP이 남았다. MAP 기능을 이용하여 개의 SNP를 20개의 염색체로 분류하고, SNP 간 재조합 정도에 따라 유전적 거리를 계산했다. 역병 저항성 검정 표현형과 SNP 유전자형 간 통계적 연관성은 BIP 기능의 ICIM (Inclusive composite interval mapping)-ADD 옵션으로 수행했다. 통계적 유의성(log of odds, LOD)에 대한 기준(LOD threshold)은 1,000번의 permutation test (p < 0.05)로 결정되었다. 통계적으로 유의한 SNP들로부터 추정된 상가적 효과(additive effect)는 유묘의 고사율(%)에 대한 천알 대비 대풍 대립유전자의 효과로 나타냈다. 상가적 효과는 각 SNP에 대해 대풍 대립유전자를 가지는 계통의 표현형(유묘의 고사율%) 평균에서 천알 대립유전자를 가지는 계통의 표현형 평균 간 차이를 대립유전자의 개수 2로 나눈 값이다.
결과 및 고찰
대풍 × 천알 재조합자식 계통 집단의 표현형 분리
균주40468에 대한 저항성 반응 검정 평가 결과 모본인 대풍은 감수성을 보였고, 부본인 천알은 저항성을 나타냈다(Fig. 1). 대풍 × 천알 재조합자식 계통 38개 중에서는 저항성과 감수성 계통은 각각 18개로 동일했고, 2개 계통은 반복적으로 40~60% 고사율을 보임에 따라 중간형으로 판단했다(Table 1). F6 파생 재조합자식 계통 38개에서 기대되는 저항성, 중간형, 감수성의 분리비는 18.4:1.2:18.4로, 이 분리비에 대해 적합도 검정(goodness-of-fit test)을 수행한 결과, 관찰된 분리비가 기대된 분리비와 유의미한 차이가 없었다(Table 2; χ2 = 0.57, p = 0.75). 이 결과는 이 집단에서 관찰된 역병 반응이 단일유전자에 의한 표현형 분리임을 함축한다.
Table 1.
Goodness-of-fit test for the segregation ratios of 38 recombinant inbred lines (RILs) of the Daepung × CheonAl population following inoculation with P. sojae isolate KACC.40468.
| Parents and RILs | Observed numbers | Expected numbers | Goodness-of-fit | |||||
| R | I | S | R | I | S | χ2 | p | |
| Daepung (P1) | 8 | |||||||
| CheonAl (P2) | 8 | |||||||
| Daepung × CheonAl (RILs) | 18 | 2 | 18 | 18.4 | 1.2 | 18.4 | 0.57 | 0.75 |
Table 2.
Fifty-two single nucleotide polymorphisms (SNPs) that were significantly associated with resistance to P. sojae isolate KACC.40468.
| Chra |
Position (bp)b | SNP ID | Daepung (S) | CheonAl (R) |
Adjusted p-valuec | R2d | ||
| Allele | Frequency | Allele | Frequency | |||||
| 18 | 55,912,198 | AX-90455222 | G | 0.49 | C | 0.51 | 1.0E-21 | 0.96 |
| 18 | 55,912,849 | AX-90485402 | C | 0.49 | T | 0.51 | 1.0E-21 | 0.96 |
| 18 | 55,924,093 | AX-90482670 | T | 0.49 | C | 0.51 | 1.0E-21 | 0.96 |
| 18 | 55,944,116 | AX-90490662 | T | 0.50 | G | 0.50 | 3.5E-26 | 0.98 |
| 18 | 55,950,341 | AX-90506324 | T | 0.50 | A | 0.50 | 3.5E-26 | 0.98 |
| 18 | 55,952,375 | AX-90466113 | C | 0.49 | A | 0.51 | 1.0E-21 | 0.96 |
| 18 | 55,954,722 | AX-90456267 | C | 0.49 | A | 0.51 | 1.0E-21 | 0.96 |
| 18 | 55,957,365 | AX-90493801 | A | 0.49 | C | 0.51 | 1.0E-21 | 0.96 |
| 18 | 55,965,452 | AX-90456611 | C | 0.49 | A | 0.51 | 1.0E-21 | 0.96 |
| 18 | 55,975,366 | AX-90334976 | G | 0.49 | A | 0.51 | 1.0E-21 | 0.96 |
| 18 | 55,983,605 | AX-90402411 | T | 0.50 | C | 0.50 | 3.5E-26 | 0.98 |
| 18 | 55,983,683 | AX-90409231 | A | 0.50 | C | 0.50 | 3.5E-26 | 0.98 |
| 18 | 55,984,097 | AX-90363270 | G | 0.50 | A | 0.50 | 3.5E-26 | 0.98 |
| 18 | 55,994,749 | AX-90435459 | T | 0.50 | C | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,012,340 | AX-90305752 | A | 0.49 | G | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,023,959 | AX-90419372 | A | 0.49 | G | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,033,039 | AX-90388943 | C | 0.49 | A | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,037,382 | AX-90400765 | G | 0.49 | A | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,037,748 | AX-90454243 | C | 0.50 | A | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,038,945 | AX-90473794 | G | 0.49 | A | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,040,302 | AX-90317341 | T | 0.50 | G | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,040,608 | AX-90444242 | T | 0.50 | C | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,040,898 | AX-90512470 | T | 0.50 | C | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,056,526 | AX-90445535 | T | 0.49 | C | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,058,777 | AX-90361126 | C | 0.50 | T | 0.50 | 7.9E-21 | 0.96 |
| 18 | 56,065,802 | AX-90511648 | T | 0.49 | C | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,075,982 | AX-90487878 | C | 0.50 | T | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,077,545 | AX-90391396 | T | 0.49 | G | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,078,131 | AX-90339253 | C | 0.50 | G | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,102,697 | AX-90476841 | G | 0.49 | A | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,123,124 | AX-90413289 | T | 0.50 | C | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,127,446 | AX-90456433 | G | 0.49 | T | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,127,800 | AX-90386532 | C | 0.49 | A | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,130,758 | AX-90360889 | A | 0.49 | G | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,144,514 | AX-90344000 | C | 0.49 | A | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,144,997 | AX-90462358 | G | 0.49 | A | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,145,524 | AX-90457110 | C | 0.49 | T | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,187,634 | AX-90413871 | C | 0.50 | T | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,187,903 | AX-90461873 | A | 0.50 | G | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,201,329 | AX-90378479 | T | 0.47 | A | 0.53 | 1.0E-15 | 0.91 |
| 18 | 56,214,870 | AX-90448445 | C | 0.50 | T | 0.50 | 7.9E-21 | 0.96 |
| 18 | 56,230,603 | AX-90512807 | C | 0.50 | T | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,232,280 | AX-90374236 | C | 0.50 | T | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,278,301 | AX-90453819 | T | 0.49 | C | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,279,910 | AX-90370238 | T | 0.50 | G | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,280,800 | AX-90417313 | C | 0.50 | T | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,281,354 | AX-90483014 | G | 0.50 | C | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,299,436 | AX-90516634 | T | 0.50 | C | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,321,318 | AX-90428430 | G | 0.50 | A | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,346,353 | AX-90475392 | A | 0.49 | C | 0.51 | 1.0E-21 | 0.96 |
| 18 | 56,353,723 | AX-90370966 | A | 0.50 | G | 0.50 | 3.5E-26 | 0.98 |
| 18 | 56,370,352 | AX-90510781 | T | 0.39 | C | 0.61 | 8.6E-13 | 0.90 |
bPhysical positions are based on the soybean genome Glyma2 (http://soybase.org).
SNP 유전자형에 대한 일원분산분석(single-factor ANOVA)
표현형과 SNP 유전자형의 연관을 확인하기 위해, 각 SNP에 대해 일원분산분석을 수행했다. 각 SNP의 대립유전자형에 따라 구분된 계통들 그룹간 평균 고사율(%)이 유의미한 차이를 보이는지 분산분석법을 통해 비교했다. 총 22,637개 SNP에 대한 분석 결과, 염색체 18번에서 다수의 SNP이 p < 4.4E-7보다 낮아 유의했고, R2를 값을 함께 고려할 때, 55.9-56.4 megabase pairs (Mbp) 지역에 위치한 52개의 SNP이 통계적으로 가장 유의하면서 높은 R2값을 가졌다(p < 1.0E-15, R2> 0.9) (Table 2). 이 범위 내에서도 다수의 SNP이 1에 근접한 결정계수(R2 = ~0.98)를 가지는 것으로 볼 때, 이 유전자좌에 역병 균주40468에 대한 천알의 저항성 유전자가 존재할 가능성이 매우 높다.
대풍 × 천알 집단의 유전자 지도 작성
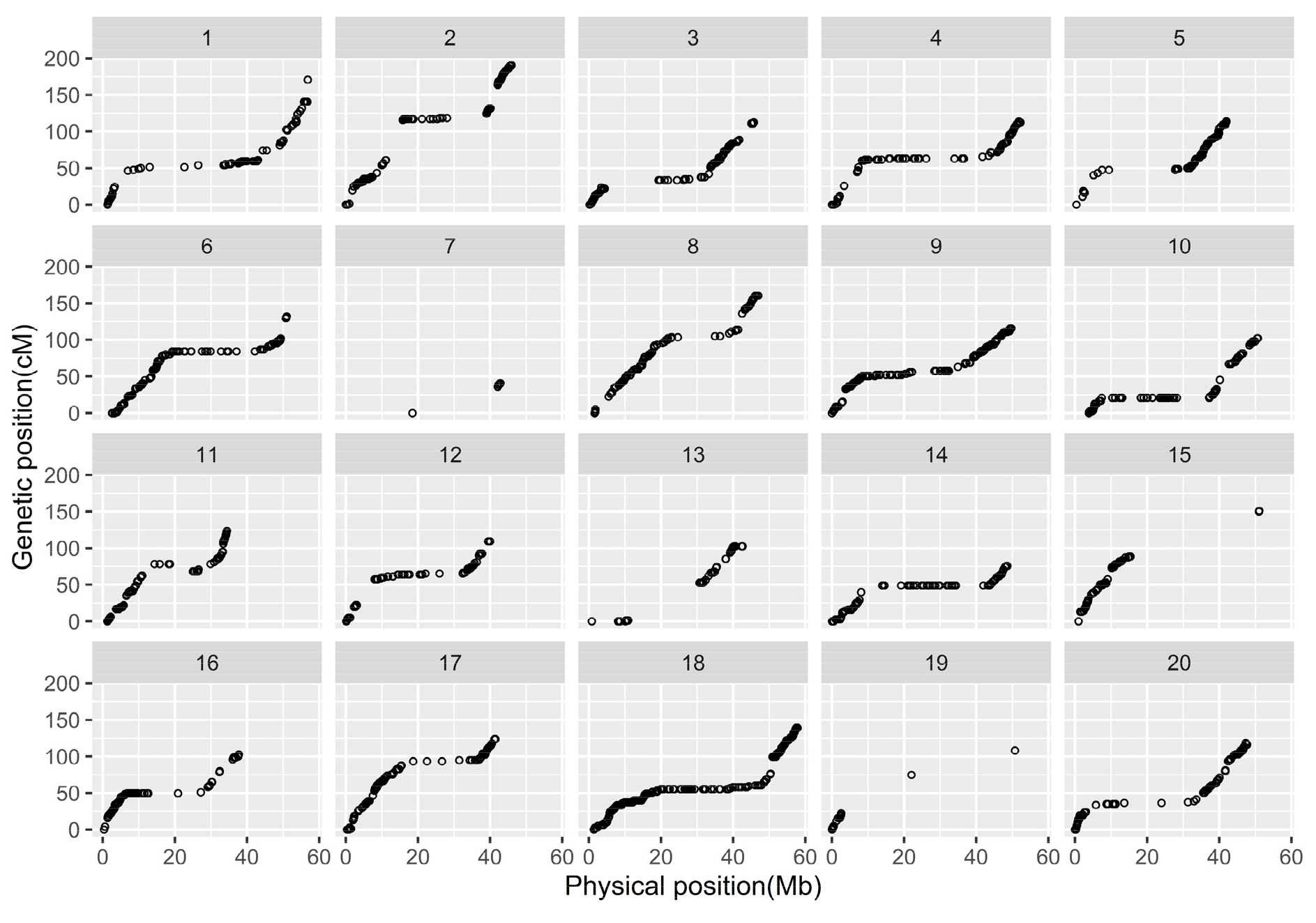
대풍 × 천알 집단의 균주40468에 대한 저항성 연관 유전자 위치를 확인하고자 고밀도 유전자 지도를 작성했고, 이 지도에 대한 세부 정보는 Table 3에 요약했다. Binning을 통해 추려진 2,513개 SNP 중 2,322개가 20개 염색체를 형성했다. 각 염색체의 유전자 지도에 통합된 SNP들의 물리적 위치와 유전적 거리 관계를 비교할 때, 중심체 주변 지역(pericentromeric region)에서는 물리적 위치의 변화에 따라 유전적 위치가 변하지 않고, 염색체의 양 말단에서는 정의 상관 관계를 보여 이론적으로 매우 바람직한 유전자 지도가 작성되었다(Fig. 2). 집단의 크기가 작아 일부 마커 간 co-segregation 되는 경향이 높았다. 유전자 지도 상에 존재하는 마커간 구간(bin)의 개수는 785개였고, 전체 길이 2,407 cM을 bin의 개수로 나누어서 계산한 마커 간 평균거리는 3.1 cM이다. 염색체 7, 19번을 제외하고 나머지 18개 염색체에서는 평균 간격이 2.0~5.7 cM로 SNP의 분포가 비교적 균일한 유전자 지도를 작성하였다(Table 3). 염색체 18번은 가장 많은 70개 구간(bin)이 형성되었고, 구간 평균 간격이 2.0 cM로 가장 가까웠다. 종합적으로, 집단 크기의 한계로 인해 사용된 SNP의 개수에 비해 고 해상도(high resolution)의 유전자 지도는 아니지만, 단일유전자에 의한 형질에 대한 분석을 수행하기에는 충분히 양질의 유전자 지도를 작성했다(Fig. 2, Table 3).
예외적으로, 염색체 7, 19번의 경우 SNP 칩 데이터로부터 8,126개와 8,166개 SNP에 대한 결과를 얻었으나 전 처리 과정에서 다형성을 보인 SNP의 수는 각각 26개, 86개뿐이었다. 결과적으로 유전자 지도가 포함하는 염색체의 영역이 특정 지역으로 한정되고 SNP 간 평균 간격도 8.2, 9.0 cM으로 타 염색체에 비해 2~4배 멀었다(Fig. 2, Table 3). 두 염색체에서 다형성을 보이는 SNP의 수가 특이적으로 적은 것(≤1%)으로 보아 이 두 염색체에서 대풍과 천알의 유전체가 상당히 유사하다고 판단된다. 대풍의 육성계보를 보면, 부모 세대에서 신팔달2호가 교배에 사용되었고, 천알의 모부본 중 하나는 SS2-2이다. SS2-2는 신팔달2호에 EMS (Ethyl-methanesulfonate)를 처리해서 얻은 포인트 돌연변이(point mutation) 계통이므로, 신팔달2호와 SS2-2가 전체 유전체 상 유사도는 매우 높을 것이다. 선행 연구에서, SS2-2와 신팔달2호 간의 다형성을 보이는 마커를 선발하고자 서로 다른 연관군(linkage group)에 위치한 SSR 마커 10개를 선발하여 SS2-2와 신팔달2호에 테스트한 결과, 1개 SSR 마커만 다형성을 보였다(Van et al., 2003). 따라서, 특정 염색체에서 대풍과 천알 간 다형성이 극히 적은 이유는 이 두 계통을 육성 과정에서 사용된 육종 재료 때문인 것으로 판단된다.
Table 3.
Information about the genetic map of the Daepung × CheonAl population.
| Chra |
Length of the map (cMb) (x) |
No. of polymorphic SNP |
No. of SNP after binning |
No. of mapped SNPc |
No. of SNP bin (y)d |
Avg. distance between SNP (cMb) (x/y) |
| 1 | 171 | 836 | 101 | 99 | 39 | 4.4 |
| 2 | 191 | 1,699 | 156 | 132 | 44 | 4.3 |
| 3 | 112 | 1,110 | 133 | 113 | 45 | 2.5 |
| 4 | 114 | 1,337 | 142 | 122 | 41 | 2.8 |
| 5 | 114 | 1,072 | 109 | 107 | 44 | 2.6 |
| 6 | 132 | 1,577 | 164 | 152 | 48 | 2.8 |
| 7e | 41 | 26 | 10 | 10 | 5 | 8.2 |
| 8 | 160 | 1,249 | 127 | 124 | 62 | 2.6 |
| 9 | 116 | 2,097 | 183 | 173 | 54 | 2.1 |
| 10 | 102 | 1,250 | 124 | 118 | 34 | 3.0 |
| 11 | 124 | 832 | 127 | 108 | 43 | 2.9 |
| 12 | 109 | 1,000 | 108 | 102 | 25 | 4.4 |
| 13 | 102 | 837 | 82 | 56 | 18 | 5.7 |
| 14 | 76 | 1,277 | 127 | 123 | 30 | 2.5 |
| 15 | 151 | 959 | 118 | 102 | 34 | 4.5 |
| 16 | 103 | 983 | 109 | 107 | 33 | 3.1 |
| 17 | 124 | 954 | 143 | 140 | 55 | 2.2 |
| 18 | 139 | 2,557 | 303 | 289 | 70 | 2.0 |
| 19d | 108 | 86 | 20 | 20 | 12 | 9.0 |
| 20 | 119 | 899 | 127 | 125 | 49 | 2.4 |
| Total | 2,407 | 22,637 | 2,513 | 2,322 | 785 | 3.1 |
유전자지도를 이용한 linkage analysis
유전자지도를 이용하여 Linkage analysis를 수행하여 역병 균주40468에 대한 저항성 유전자좌를 찾았다(Table 4). 염색체 18번 55.9-56.4 Mbp 지역에서 가장 유의미한 LOD 값(LOD = 23.1)을 가졌고, 이 유전자좌가 설명하는 표현형 분산은 93.9%이다. 대풍의 대립유전자의 상가적 효과(additive effect)는 47.4로서 이는 대립유전자 1개가 천알에서 대풍으로 대체될 때 유묘의 고사율(%)이 47.4% 포인트 높아짐(감수성)을 의미하고 반대로 대풍에서 천알로 대체되는 경우에는 고사율이 47.4% 포인트 낮아짐(저항성)을 의미한다(Table 4).
Table 4.
The genomic region identified for resistance to P. sojae isolate KACC.40468 in the Daepung × CheonAl RIL population.
| Chra | Position (bp)b | Flanking markers of interval | LODc | PVE (%)d | ae |
| 18 |
55,912,198 to 56,412,565 |
AX-90455222 to AX-90366982 | 23.1 | 93.9 | 47.4 |
Linkage analysis를 통해 특정된 유전자좌의 범위는 약 0.5 Mbp (55.9~56.4 Mbp)로서, 분리 집단의 작은 규모로 인해 유전자 지도의 해상도(resolution)을 높이는 데는 한계가 있었다. 선행연구에 따르면 염색체 18번의 해당 지역에서는 최소 5개의 Rps 유전자가 보고되었다; Rps4 (Athow et al., 1980; Sandhu et al., 2004), Rps5 (Buzzell & Anderson, 1981), Rps6 (Athow, 1982; Gordon et al., 2007), Rps12 (Sahoo et al., 2017), RpsJS (Sun et al., 2014), Rps13 (Sahoo et al., 2021). 기존에 보고된 저항성 연관 분자 마커를 기반으로 Rps4 (54.5-56.3 Mb), Rps5 (53.9 Mb), Rps6 (54.5 Mb), Rps12 (56-56.3 Mb), RpsJS (56.3-56.6 Mb)의 물리적 위치를 본 연구에서 찾은 천알의 저항성 유전자좌 범위를 비교하였다(Fig. 3). 천알의 저항성 유전자좌 추정 범위는 비교적 넓어 RpsJS, Rps12, Rps4와 모두 겹친다. Sahoo et al. (2017)은 Rps4/6과 Rps12를 포함하는 PI 399036와 AR2의 교배로 얻은 F7 RIL 집단과 F2 개체들을 이용해 표현형 검정 및 분자 마커 검정을 수행했고, 두 유전자 Rps12와 Rps4가 집단 내에서 분리된다는 것을 보임으로써 이들이 인접한 서로 다른 유전자임을 밝혔다. 천알과 타 품종과의 유전자 간 관계를 확인하기 위해서는 동일한 어프로치가 필요하고, 현 결과로는 천알의 저항성 유전자가 Rps4 또는 Rps12와 동일한 유전자의 대립유전자형(alternative allele)일 가능성과 이와는 다른 새로운 Rps 유전자일 가능성이 모두 존재한다.
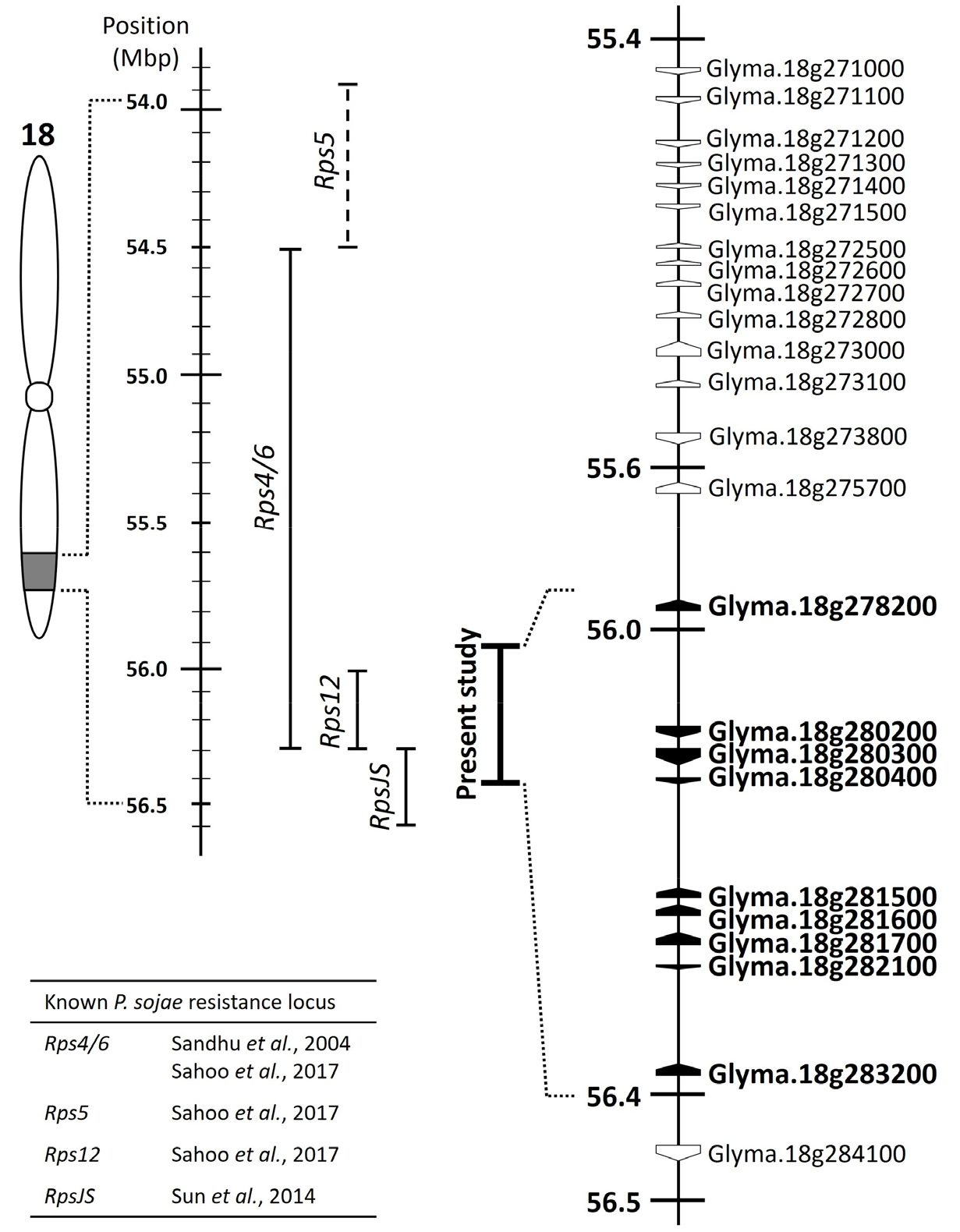
Fig. 3.
A schematic diagram of the resistance locus identified in chromosome 18. Physical map, known Rps genes, and the annotated genes encoding leucine-rich repeat and serine/threonine kinase proteins are presented, based on the Glyma.Wm82.a2.v1. The predicted genes in the target interval are highlighted in bold.
천알의 역병 저항성 유전자좌 발견의 의의
천알은 국내 품종(계통) 중에서는 역병저항성 유전자좌가 보고되는 3번째 품종이고 그 대상인 균주40468를 기준으로는 처음 보고되는 저항성 유전자좌이다. 선행 연구에서 ‘청자’와 ‘풍산나물’이 균주40468 (a.k.a. P-98145)에 대해 저항성을 보였다(Kang et al., 2019). 청자의 역병 저항성에 대해서는 유전집단을 이용한 연구가 진행중이다. 풍산나물에 대해서는 아직 연구되지 않았지만 천알이 ‘풍산나물’ × ‘SS2-2’ 교배집단에서 선발된 계통이고 SS2-2가 균주40468에 대해 감수성인 것으로 보아(Supplementary Fig. 1) 천알의 저항성 유전자좌는 풍산나물로부터 유래된 대립유전자(allele)일 것으로 판단된다.
현재까지 국내 콩 품종 중에서는 대원과 소청2호에서 각각 1개(균주2457), 2개(균주40412 & 균주2457) 균주에 대해서 역병 저항성 유전자좌가 보고되었다(Jang et al., 2020a, 2020b). 흥미롭게도, 두 저항성 품종(대원, 소청2호)이 동일하게 균주2457에 대해 저항성을 보였으나 저항성 유전자좌는 서로 다른 염색체에 위치했고, 소청2호는 균주40412과 균주2457에 대해 각각 다른 염색체에 위치한 유전자좌가 저항성을 부여하는 것으로 나타났다(Jang et al., 2020a, 2020b). 균주2457에 대한 소청2호의 저항성 유전자좌로 한 염색체(18번)의 멀리 떨어진 두 지역(2.1-2.6 & 53.1-53.3 Mbp)이 통계적으로 유의성을 보였다(Jang et al., 2020b). 이는 각 품종이 가지고 있는 저항성 유전자가 여러 개일 수 있고 상대 균주에 따라 다른 저항성 유전자가 작용하기 때문에, 콩 품종-역병균주 간 상호작용(arm-race)이 매우 다양할 수 있음을 보여주는 좋은 예이다. 장기적으로 이용될 수 있는 콩 역병 저항성 품종을 육성하기 위해서는 저항성 유전자들을 집적함으로써 복수의 균주에 저항할 수 있는 중간모본을 만드는 일이 선행되어야 한다.
Linkage analysis에서 찾은 염색체 18번의 약 0.5 Mbp (55.9-56.4 Mbp) 내에 존재하는 유전자들에 대해 콩(cv. Williams82) 표준 유전체 유전자 주석(gene annotation) 정보(Glyma.Wm82.a2.v1)를 기반으로 해당 범위에 분포하는 유전자의 종류에 대해 분석하였다. 총 63개의 유전자가 존재하고 이들 중 9개가 유전자가 병 저항성 기능을 하는 leucin rich repeat (LRR) domain 또는 serine/threonine kinase (STK)를 합성하는 R-유전자인 점으로 보아 해당 유전자좌는 LRR 유전자가 반복적으로 위치한 클러스터(cluster)라고 할 수 있다(Fig. 3). 유전체 범위를 확장해서 보면, 55.4~56.5 Mbp 지역에서 총 24개의 LRR 합성 유전자들이 배열되어 있다(Fig. 3). 원핵생물의 유전체에서는 하나의 조절에 관여하는 다수의 유전자들이 인접해서 존재하는 경우들이 종종 있다. 진핵생물의 유전체에서는 이런 현상이 드물지만, nucleotide-binding (NB) LRR을 합성하는 R-유전자들이 함께 모여 있는 것은 여러 식물 유전체에서 공통적인 특성 중 하나이다(Van Wersch & Li, 2019). 이 유전자군이 생산하는 수용체(receptor) 단백질은 식물 세포막에서 세포 외부에서 발생하는 병원균의 감염 징후를 인지하는 직․간접적인 센서로 이용되고 이에 식물의 방어(defense)에서 매우 중요한 의미를 가진다. 인접한 위치에 방어 관련 유전자의 copy의 수가 많고 이들이 함께 발현되어 단백질로 번역된다면 더 효율적인 방어가 가능할 것으로 여겨진다(Van Wersch & Li, 2019). 진화적 측면에서는 유전체 상에서 이러한 R 유전자의 copy number와 식물 면역 능력 간 관계도 매우 연관성이 높다고 보고되어 있다(Tamborski & Krasileva, 2020). 따라서, 이 지역 내에서 존재하고 있는 역병 저항성 유전자를 보다 정확하게 특정하고 이 유전자에 의한 저항성 작용원리를 연구하는 것은 흥미로운 후속 연구가 될 것이다.
결론적으로, 본 연구에서는 천알을 균주40468에 대한 또 다른 저항성 자원으로 확인하였고, 저항성 유전자좌를 염색체 18번의 55.9-56.4 Mbp 범위로 특정했다. 이 결과는 기존에 연구에 대해 새로운 역병 저항성 콩 품종 개발 연구를 수행함에 있어 기초자료를 제공할 수 있을 것으로 여겨진다. 더 나아가서, 후속 연구를 통해 저항성 유전자좌의 대립유전자적 관계성(allelic relationship)이나 타겟 지역의 R 유전자의 저항성 작용 원리 등 향후 콩 역병 저항성 연구를 위한 기초 자료로 활용될 수 있을 것으로 기대된다.
적 요
콩 역병(Phytophthora root rot, PRR)은 난균(oomycete)인 Phytophthora sojae에 의해 발생하는 콩의 주요 병 중 하나로, 배수가 잘 안 되는 밭이나 습한 토양에서 심하게 발생한다. 역병의 피해를 효과적으로 줄일 수 있는 방법은 주로 역병 저항성 품종을 재배하는 것으로, 이는 저항성 유전자 Rps (resistance to P. sojae)에 대한 연구를 중심으로 이루어진다. 본 연구는 대풍 과 천알(계통명 SS0404-T5-76)을 교배하여 구축한 RIL (recombinant inbred line) 집단을 이용하여 콩 역병 균주40468과 연관된 저항성 유전자좌를 탐색하기 위해 수행되었다. 역병 균주40468에 대한 저항성 평가는 하배축 접종(hypocotyl inoculation) 방법으로 이루어졌다. 저항성 검정 결과, 천알은 저항성,대풍은 감수성을 보였고 집단 내에서는 계통들의 표현형이 분리되는 양상을 보였다. 집단 내에서 표현형 분포는 1:1 (R:S) (χ2 = 0.57, p = 0.75) 분리비와 일치하였으며, 이는 저항성 반응이 단일 유전자에 의해 조절됨을 나타낸다. 대풍, 천알과 각 RIL 계통들은 고밀도 SNP 유전자형 분석을 통해 데이터를 얻었고, 이를 바탕으로 유전자 지도를 작성하였다. 일원분산분석(Single-marker ANOVA) 및 linkage analysis 결과, 18번 염색체의 55.9~56.4 Mbp에서 높은 통계적 유의성을 보였으며, 이 지역의 표현형 분산은 ~98%로 나타났다. 탐색된 영역은 다수의 선행연구에서 Rps의 위치로 보고된 지역과 겹치며, 콩 표준 유전체 정보를 기반으로 0.5 Mbp 범위 내에서 leucine-rich repeat (LRR) 또는 serine/threonine kinase (STK)을 합성하는 유전자 9개를 포함하고 있다. 천알은 역병 균주40468에 대한 저항성 유전자좌가 밝혀진 첫 국내 콩 품종으로, 본 연구에서 밝힌 천알의 저항성 유전자좌는 향후 역병 저항성 육종 및 연구에서 유용한 재료가 될 것이다.